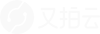

摘 要:【目的】建立双斑长跗萤叶甲成虫触角转录组数据库,筛选雌虫、雄虫触角中的差异表达基因并解析其涉及的生理功能,为进一步研究双斑长跗萤叶甲的基因功能分析及嗅觉感受机制奠定分子基础。
【方法】采用Illumina高通量测序技术对双斑长跗萤叶甲雌虫、雄虫触角进行转录组测序,对获得的unigenes进行功能注释、通路富集等生物信息学分析,利用RT-qPCR技术检测气味结合蛋白基因在触角中的表达水平。
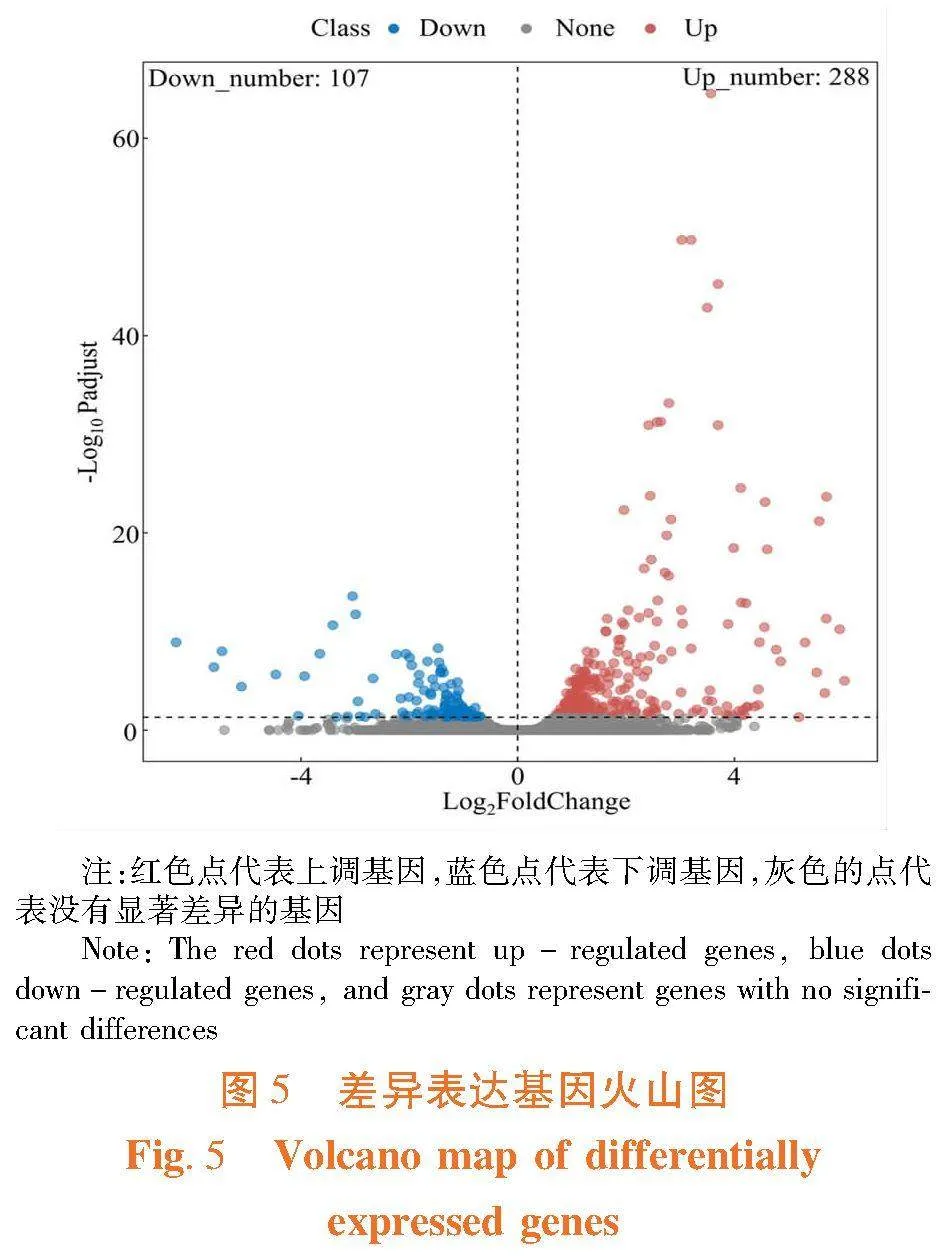
【结果】双斑长跗萤叶甲雌、雄成虫触角转录组测序共获得73 050条unigenes,Q30碱基百分比均在93.23%以上,34 233条unigenes至少在1个数据库中获得注释。与双斑长跗萤叶甲雌虫触角相比,雄虫触角中检测到395个差异表达基因,其中288个基因上调表达,107个基因下调表达。
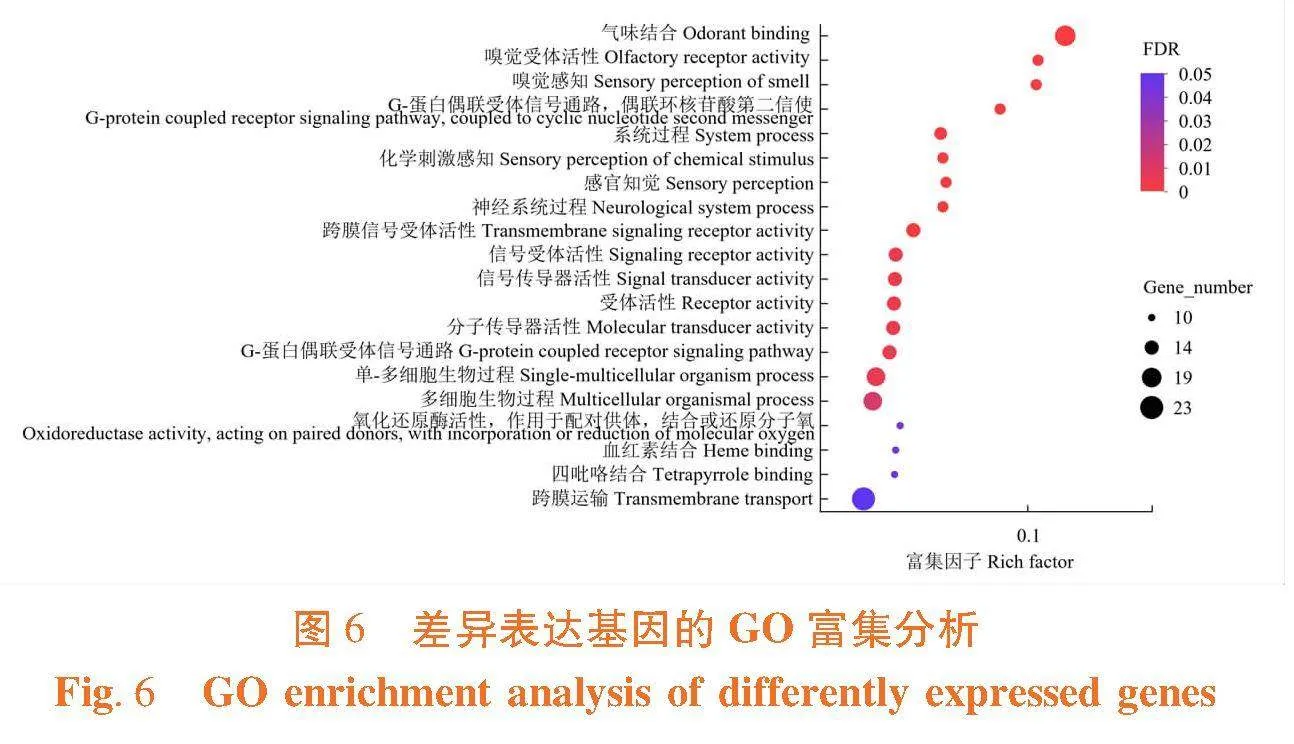
【结论】在GO注释库中,差异表达基因富集最显著的功能为气味结合、嗅觉受体活性和嗅觉感知。气味结合蛋白基因在雌虫、雄虫触角中的相对表达水平趋势与转录组测序结果相符合,获得了双斑长跗萤叶甲成虫触角转录组数据库。
关键词:双斑长跗萤叶甲;触角转录组;功能注释;差异表达基因
中图分类号:S433 文献标志码:A 文章编号:1001-4330(2024)04-0984-11
0 引 言
【研究意义】双斑长跗萤叶甲Monolepta signata,属于鞘翅目Coleoptera,叶甲科Chrysomelidae,萤叶甲亚科Galerucinae,长跗萤叶甲属Monolepta[1-3]。该叶甲在世界上主要分布于亚洲东部和东南部;在我国主要分布于西北、东南和西南地区[4-7]。双斑长跗萤叶甲为多食性昆虫,不仅为害玉米、棉花、高粱、豆类和十字花科蔬菜等多种作物,还取食多种杂草,且能在田间杂草和农作物之间转移为害[8, 9]。双斑长跗萤叶甲以幼虫为害植物根部,成虫取食寄主植物的叶片、花和果穗,具有为害期长、虫口密度高、繁殖力强、扩散快等特点,危害多种作物[10, 11]。【前人研究进展】昆虫利用灵敏的嗅觉系统来识别环境中的化学信号,并根据接收到的信号进行自身的生命活动,如定位寄主、寻找配偶和躲避天敌[15, 16]。昆虫在寻找配偶时,雄虫感受到雌虫释放的性信息素后沿着迹象寻找雌虫,接近雌虫后释放自身的信息素,雌虫除了根据雄虫释放的信息素评估后进行交配之外,交配后也要依据寄主挥发物定位产卵场所[17, 18]。触角是昆虫进行化学信号识别的主要器官,雌虫、雄虫触角基因表达水平的差异直接影响了二者对环境中化学信号的识别与接收,最终导致了雌虫、雄虫生理和行为的差异[19]。转录组测序是对细胞或组织在某一生理条件下的所有转录信息进行高通量测序分析,转录组学是对生物体所有基因的表达调控、蛋白质功能等信息进行研究[21, 22]。高通量测序技术和分子生物学技术的快速发展为转录组学研究奠定了基础[23, 24],随着新一代高通量测序技术在昆虫上的应用,目前对30余种鞘翅目昆虫进行了触角的转录组测序和分析[25-27],筛选出了参与嗅觉识别过程的相关蛋白基因,并利用生物信息学和分子生物学技术对关键基因进行了序列分析和功能验证[28-31]。【本研究切入点】目前,对双斑长跗萤叶甲的研究主要集中于生物学特性和综合防治[12-14],关于其触角在嗅觉识别过程的分子机制尚不清楚。
利用扫描电镜观察了双斑长跗萤叶甲雌成虫、雄成虫触角形态及感器类型 [20],而触角是如何执行嗅觉功能的分子机制尚未报道。需选择双斑长跗萤叶甲成虫触角进行转录组分析及雌虫、雄虫差异基因的筛选,明确双斑长跗萤叶甲成虫触角中的基因信息。【拟解决的关键问题】利用Illumina高通量测序技术对该叶甲雌、雄成虫触角进行转录组测序,构建双斑长跗萤叶甲触角转录组数据库,对获得的unigenes进行功能注释,筛选出在双斑长跗萤叶甲雌虫、雄虫触角中差异表达基因,分析双斑长跗萤叶甲雌虫、雄虫生理和行为差异的关键基因,为研究该叶甲触角的基因功能和嗅觉感受机制奠定分子基础。
1 材料与方法
1.1 材 料
双斑长跗萤叶甲成虫采集于新疆塔城地区沙湾市南湾镇东湾村玉米地(85°48′E,44°03′N),实验室内饲养,养虫室温度28℃,相对湿度控制在40%~50%,每日饲喂新鲜的棉花叶片。分别选择活动能力较强的雌成虫、雄成虫并用显微手术镊子和剪刀切取触角,以100对触角为一个样品,雌虫、雄虫各设置3个重复。将触角与虫体分离后迅速放入浸泡在液氮的离心管中,保存于-80℃低温冰箱。
1.2 方 法
1.2.1 RNA提取、cDNA文库构建及测序
用Trizol试剂(Invitrogen, USA)提取双斑长跗萤叶甲雌、雄触角样品的总RNA,RNA的质量和完整性用1.0%的琼脂糖凝胶电泳和Agilent 2100 bioanalyzer(Agilent Technologies, CA, USA)进行检测。检测合格的RNA样品由北京诺禾致源科技股份有限公司完成cDNA文库的构建与测序,测序平台为Illumina NovaSeq 6000(Illumina, USA)。利用Oligo(dt)的磁珠富集带有Poly A尾的mRNA后加入片段缓冲液,使其成为短片段mRNA,以短片段mRNA为模板,随机寡核苷酸为引物合成cDNA第一条链。在DNA polymerase I体系下,以dNTPs为原料合成cDNA第二条链,用AMPure XP 系统(Beckman Coulter, Beverly, USA)筛选修复处理后的250~300 bp的片段,用PCR扩增后再次纯化后建库并质检,质检合格后上机测序。
1.2.2 数据拼接与组装
对包含带有测序接头和测序质量较低的reads原始数据进行过滤,对测序错误率和GC含量分布进行检查,Fastp软件(version 0.19.7)被用于数据质量把控,保证获得的clean reads能够用于后续组装。用Trinity软件(version 2.6.6,默认参数)对clean reads进行拼接与组装,用BUSCO软件对拼接结果进行质量评估,确保得到高质量的转录本序列[32]。
1.2.3 基因功能注释
利用Blast软件(version 2.2.28+)将获得的双斑长跗萤叶甲触角unigenes与NCBI蛋白数据库(NCBI non-redundant protein sequences,NR)、GO数据库、KEGG数据库、Swiss-Prot数据库(A manually annotated and reviewed protein sequence database)、蛋白家族数据库(Protein family database,PFAM)、直系同源基因簇(Clusters of orthologous groups of protein,KOG/COG)和NCBI核酸序列数据库(NCBI nucleotide sequences,NT)进行比对,获得unigenes的注释信息。
1.2.4 差异表达基因
利用基于负二项分布的DESeq2软件(version 1.6.3)对双斑长跗萤叶甲雌、雄虫触角的转录组数据进行组间差异表达分析,标准化处理后计算得到Fold Change,根据Padjust lt; 0.05 且 log2FoldChange>1的条件,筛选双斑长跗萤叶甲雌虫、雄虫触角中具有表达差异的基因。Clusterprofiler软件(version 3.0.4)被用于差异表达基因的GO功能和KEGG通路富集分析,当Pvalue≤0.05时,认为差异表达基因在此GO功能或KEGG通路中显著富集。
1.2.5 气味结合蛋白基因相对表达水平
基于双斑长跗萤叶甲触角转录组unigenes功能注释结果,选择FPKM值(每千碱基转录序列中每百万个碱基测序的预期片段数)较高的气味结合蛋白(Odorant binding protein, OBP)基因进行RT-qPCR检测,分析其在雌虫、雄虫触角中的相对表达水平。选择甘油醛-3-磷酸脱氢酶(Glyceraldehyde-3-phosphate dehydrogenase,GAPDH)基因作为内参基因,利用NCBI Primer -Blast 在线网站(https:// www. ncbi. nlm. nih. gov/ tools/ primer-blast/ index. cgi)设计引物,由北京诺禾致源科技股份有限公司合成引物。利用CFX Connect荧光定量PCR检测系统(Bio-Rad,Hercules,CA,USA)对OBP基因进行测定,反应程序为:95℃预变性30 s后,95℃ 15 s和60℃ 30 s循环40次,将反应体系从65℃缓慢升温至95℃,以每秒0.5℃的升温速度扩增溶解曲线,每个样品设置3次重复。以各基因在雌虫触角中的表达量作为对照,用2-△△CT法计算各基因在雄虫触角中的相对表达量。
1.3 数据处理
利用SPSS软件(version 20.0)对试验数据进行统计分析,应用独立样本t测验法检验基因在双斑长跗萤叶甲雌虫触角和雄虫触角表达量间的差异显著性。表1
2 结果与分析
2.1 双斑长跗萤叶甲触角转录组测序统计与数据组装
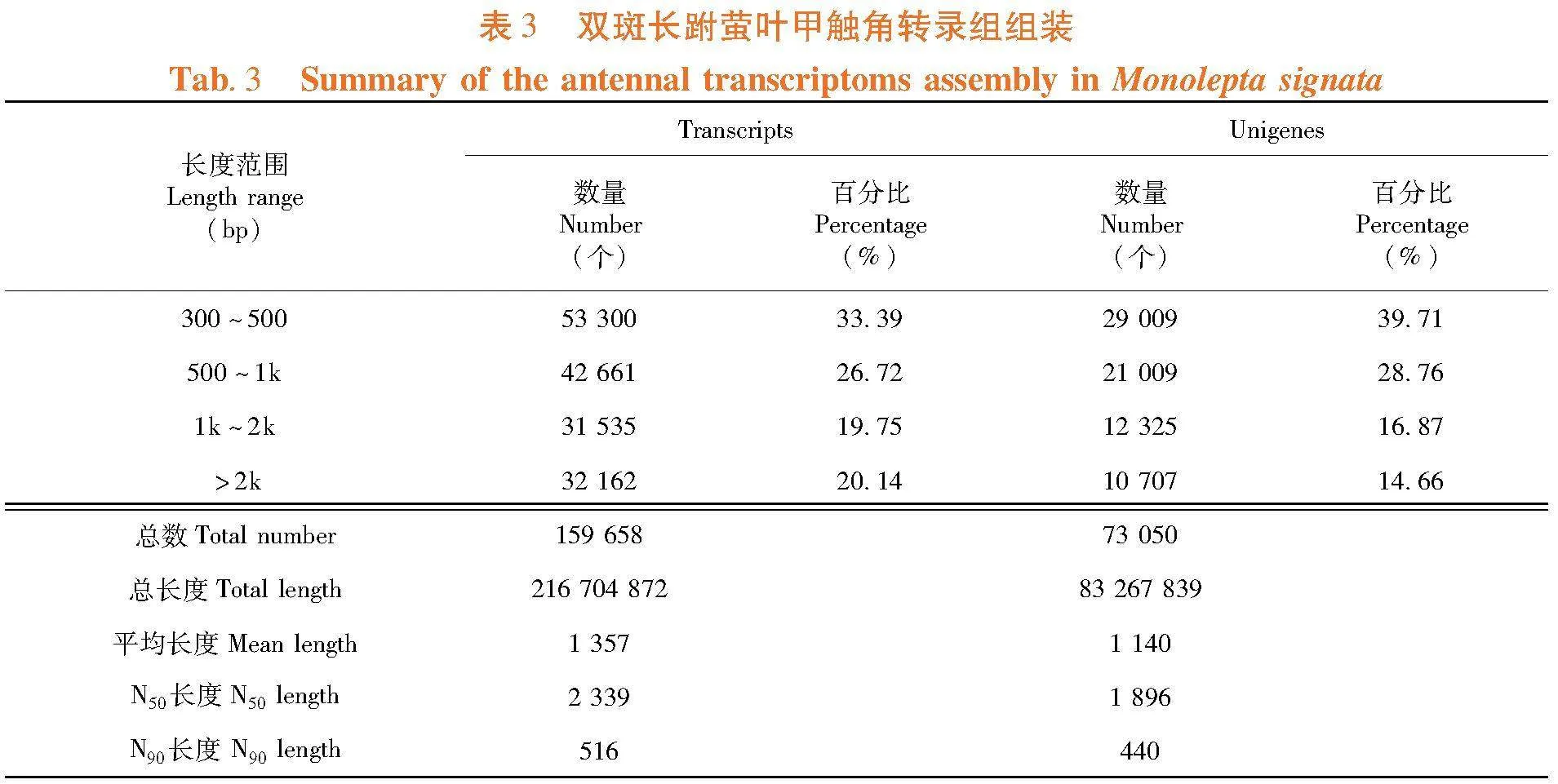
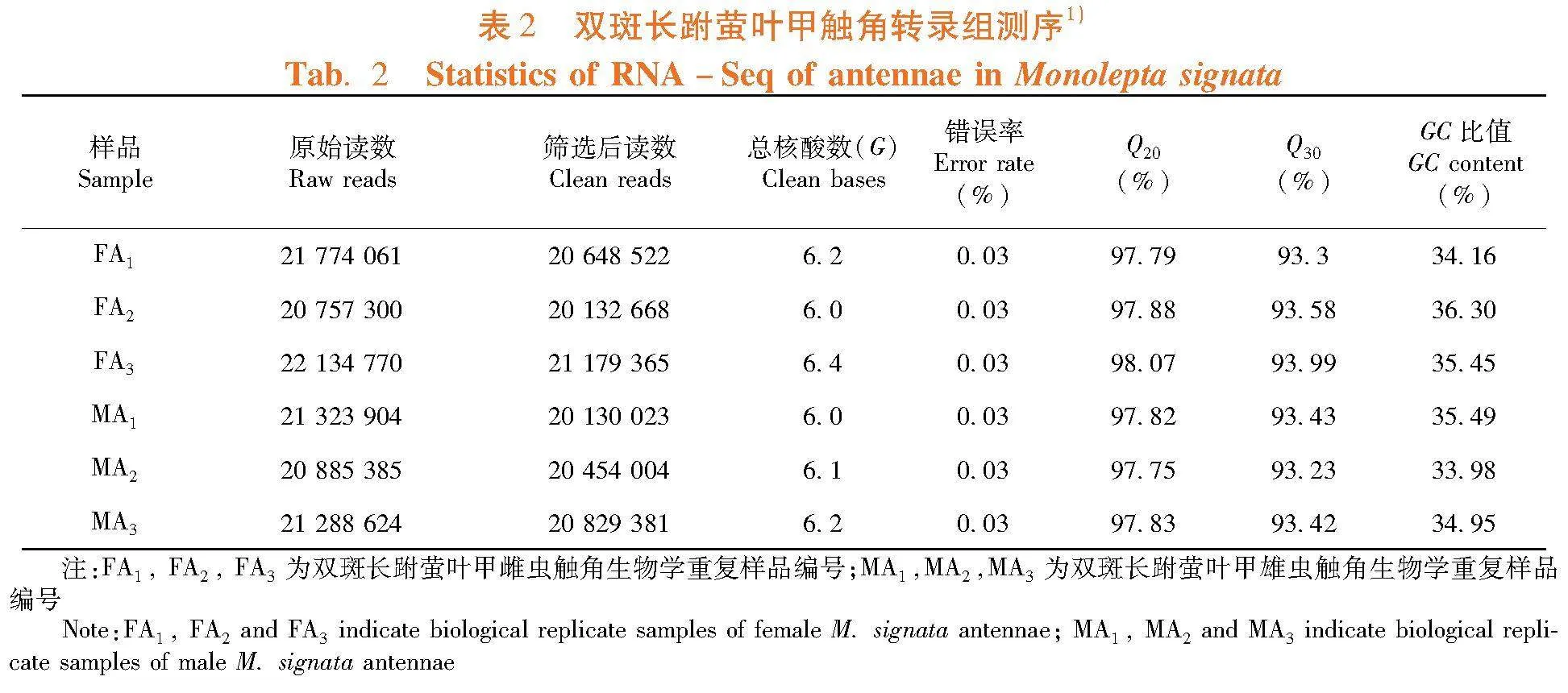
研究表明,各样品clean bases皆超过了6 Gb,GC碱基占总碱基数的百分比为35.05%。Q20和Q30所占百分比分别在97.75%和93.23%以上,clean reads的测序错误率较低,转录组测序质量较好。共得到73 050个unigenes,平均长度为1 140 bp,N50和N90的平均长度分别为1 896 bp和440 bp,unigenes长度大于1 000 bp的有23 032条,占比31.53%,组装完整性较好。转录组测序原始数据上传至NCBI的SRA(Sequence Read Archive)数据库,项目号为PRJNA960895。表2,表3
2.2 Unigenes功能注释
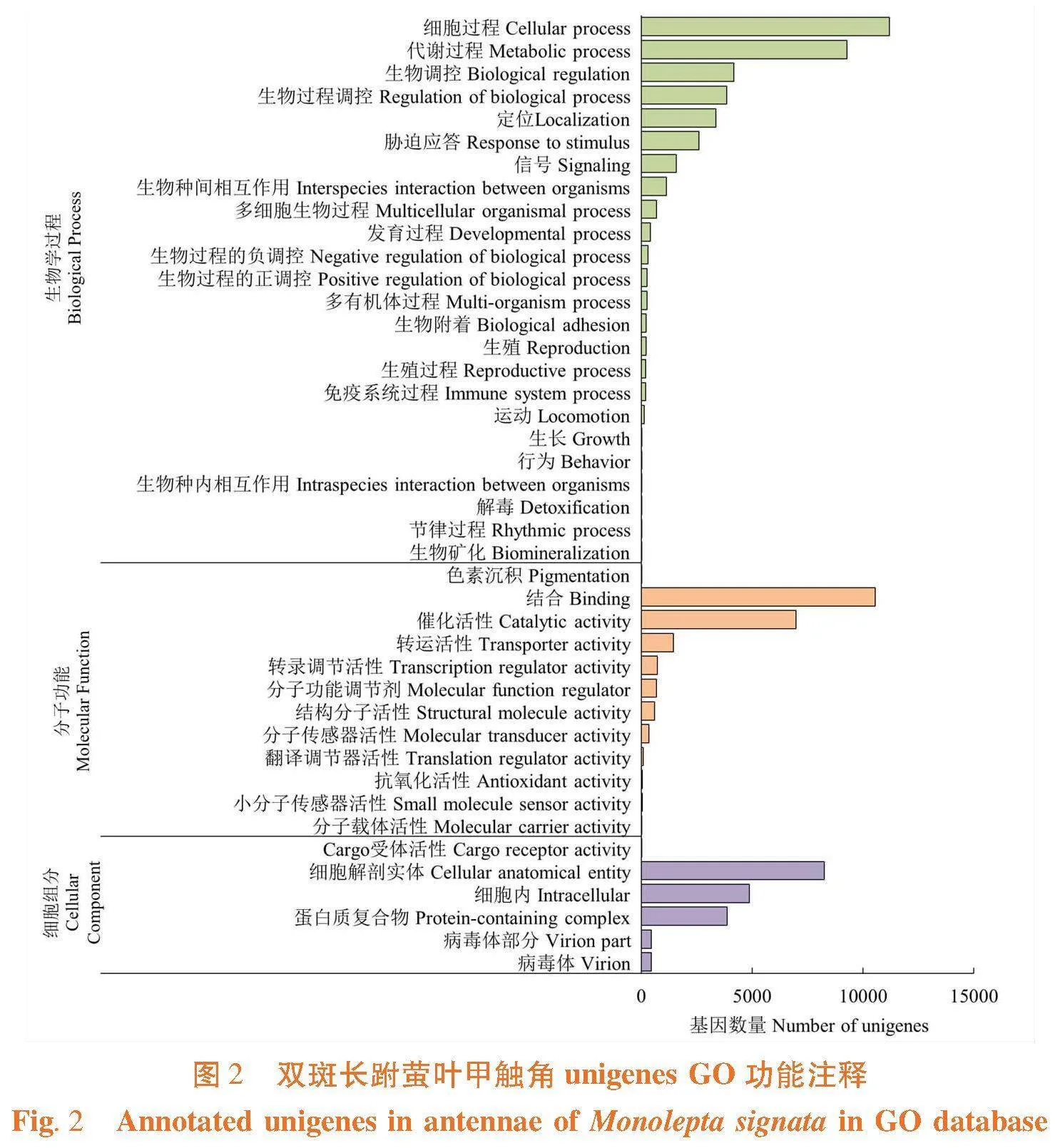
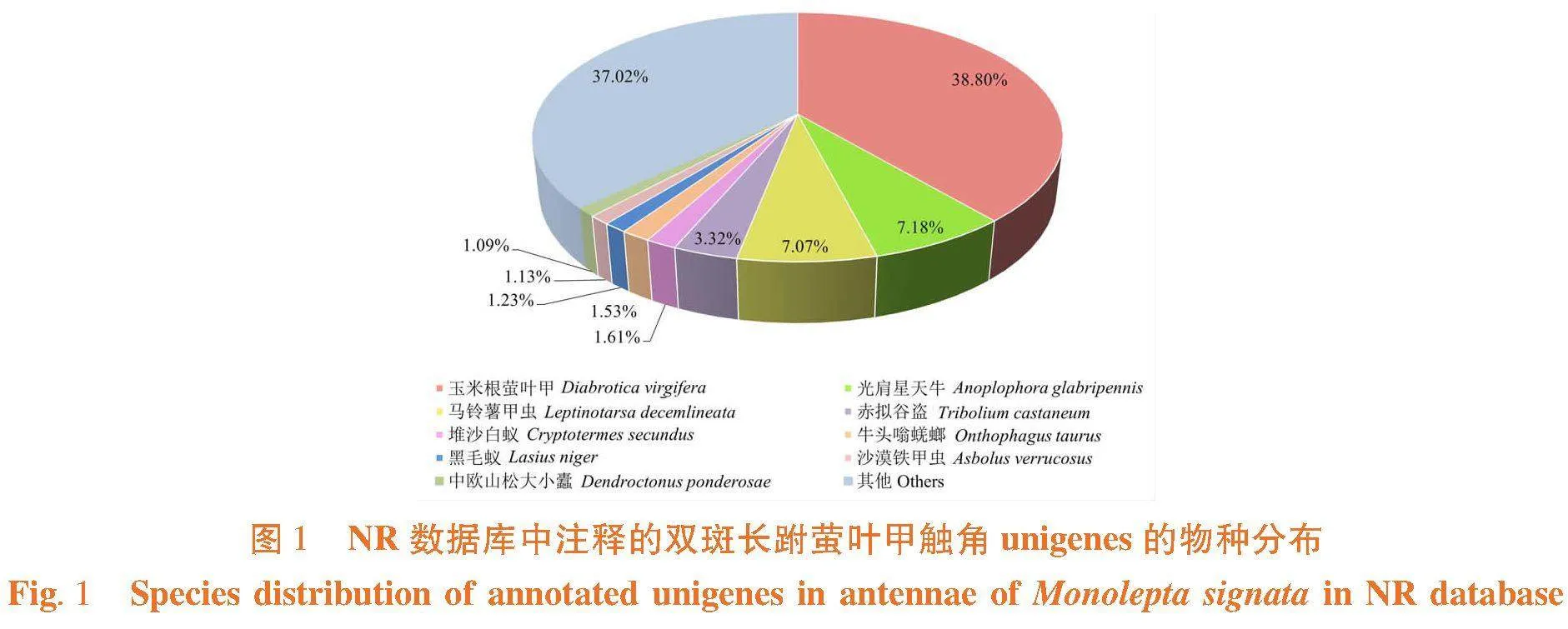
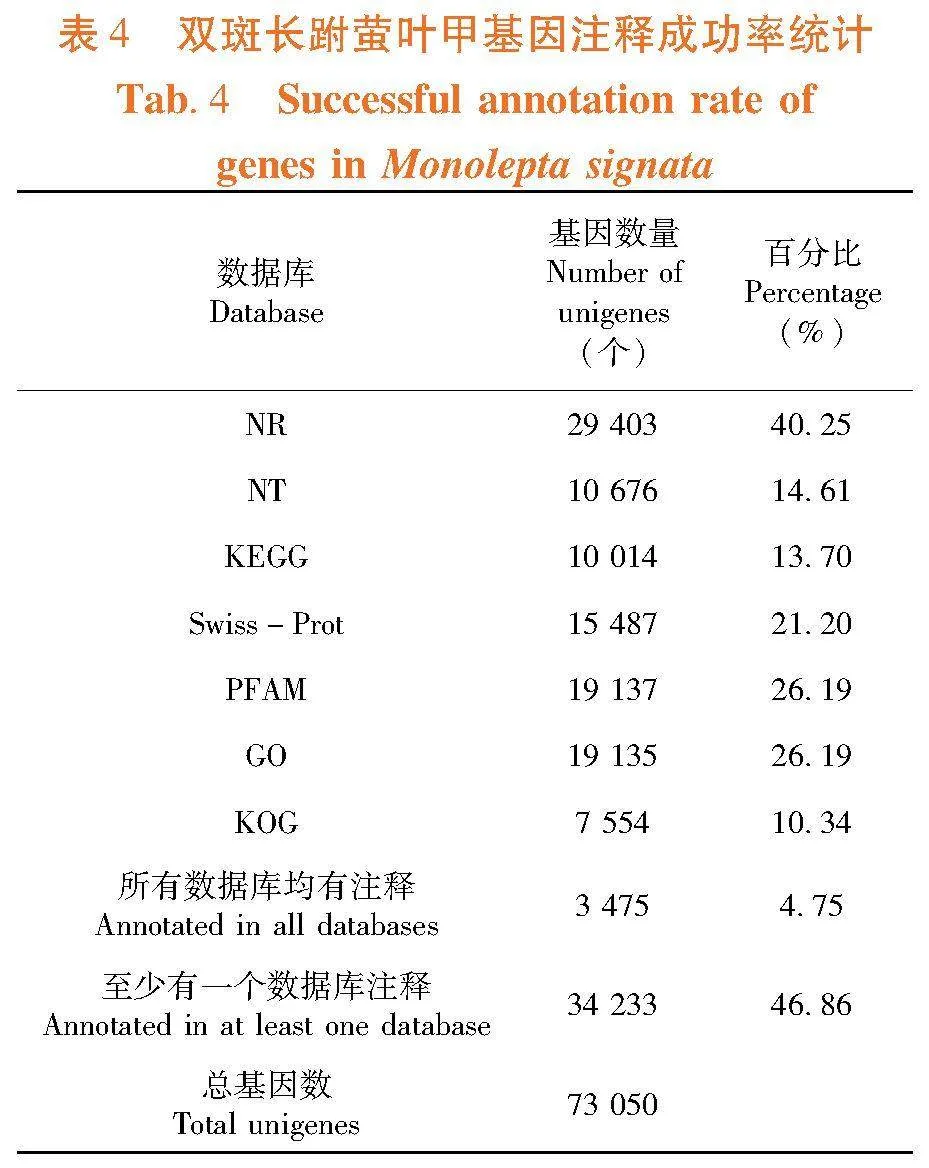
研究表明,34 233条unigenes至少在一个数据库中获得功能注释信息,占总unigenes数的46.86%。其中有3 475条unigenes在以上7个数据库中同时成功注释,占总unigenes数的4.75%。NR数据库中注释成功的unigenes数量最多,为29 403个(40.25%),其后依次是PFAM数据库为19 137个(26.19%),GO数据库为19 135个(26.19%),Swiss-Prot数据库为15 487个(21.20%),NT数据库为10 676个(14.61%),KEGG数据库为10 014个(13.70%),KOG数据库为7 554个(10.34%)。双斑长跗萤叶甲触角注释的基因序列与玉米根萤叶甲Diabrotica virgifera virgifera LeConte相似性最高,占38.80%。其次为光肩星天牛Anoplophora glabripennis(Motschulsky)(7.18%)、马铃薯甲虫Leptinotarsa decemlineata(7.07%)、赤拟谷盗Tribolium castaneum(3.32%)、堆沙白蚁Cryptotermes secundus(1.61%)、牛头嗡蜣螂Onthophagus taurus(1.53%)、黑毛蚁Lasius niger(1.23%)、沙漠铁甲虫Asbolus verrucosus(1.13%)和中欧山松大小蠹Dendroctonus ponderosae(1.09%),其他物种占37.02%。表4,图1
2.3 基因功能分类
研究表明,测序共有19 135条unigenes注释到GO数据库中,可分为生物学过程、分子功能和细胞组分3个大类,细分为42个亚类。在3大类中,unigenes注释到生物学过程的数量最多,占比50.44%;注释到分子功能这一大类的unigenes数量占比为27.06%,注释到细胞组分这一大类的unigenes数量占比最少,为22.49%。细胞过程、代谢过程和生物调节是生物学过程这一大类中unigenes数量最多的3个类别,比重分别是14.08%、11.66%和5.25%;结合和催化活性是分子功能这一大类中最主要的2个类别,unigenes数量占比分别是13.27%和8.77%;细胞解剖实体和细胞内是细胞组分中unigenes数量最多的两个类别,占比分别是10.38%和6.11%。图2
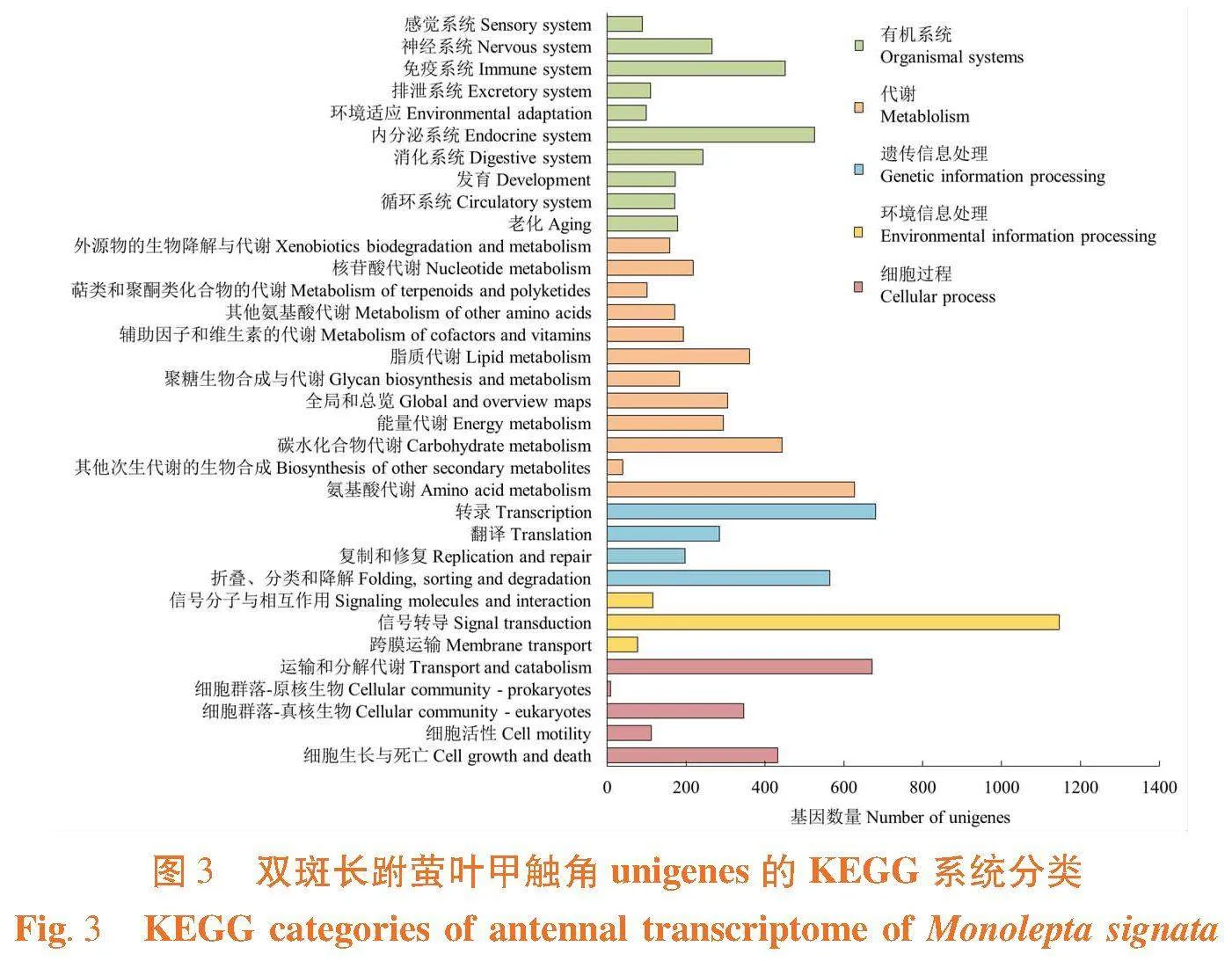
双斑长跗萤叶甲触角转录组中有10 014条unigenes在KEGG数据库中得到注释,参与了5大类代谢通路分支,即细胞过程、环境信息处理、遗传信息处理、代谢和有机系统,第二层级包括34小类,在第三层级中包括231个代谢通路。其中被注释在信号转导通路中的基因数目最多,为1 146条(11.44%),占比11.44%;其次注释unigenes数目较多的通路依次是转录681条(6.80%),运输和代谢672条(6.71%),氨基酸代谢627条(6.26%),折叠、分类和降解565条(5.64%),内分泌系统526条(5.25%),免疫系统451条(4.50%)。图3
双斑长跗萤叶甲触角转录组中的7 554条unigenes在KOG数据库中得到注释,分为25类。在R类(一般功能预测)中注释的unigenes数目最多,有1 203个,占比15.92%;其次是O类(翻译后修饰,蛋白质翻译,分子伴侣)中有881条unigenes被注释,占比1.66%;在T类(信号转导机制)有859条unigenes被注释,占比11.37%;在S类(未知功能)中有569条unigenes被注释,占比7.53%,在U类(胞内运输,分泌和囊泡运输)有496条unigenes获得注释,占比6.57%。图4
2.4 差异表达基因
研究表明,与雌虫触角相比,雄虫触角的转录组中筛选有395个差异表达基因,其中288个基因上调表达,107个基因下调表达。图5
99个上调表达基因被注释到GO的3大功能类别中,共获得806条功能注释。在生物学过程中,最多有48个基因(48.50%)注释到细胞过程,其次是39个基因(39.40%)注释到代谢过程,29个基因(29.30%)被注释到有机物代谢过程;在分子功能中,最多有48个基因(48.50%)注释到结合,其次有33个基因(33.30%)为催化活性;在细胞组分中,有27个基因(27.30%)注释为细胞膜,剩余的功能条目注释基因均不超过20个。64个下调基因被注释到GO的3大功能类别中,在生物学过程中,最多有35个基因(54.70%)注释到细胞过程,其次有27个基因(42.20%)注释到代谢过程;分子功能类别中29个基因(45.30%)注释到催化活性,27个基因(42.20%)注释到结合;细胞组分中有23个基因(35.90%)注释到细胞膜,剩余的功能条目注释基因均不超过20个。选择富集最显著的20个GO term在气泡图中展示。图6
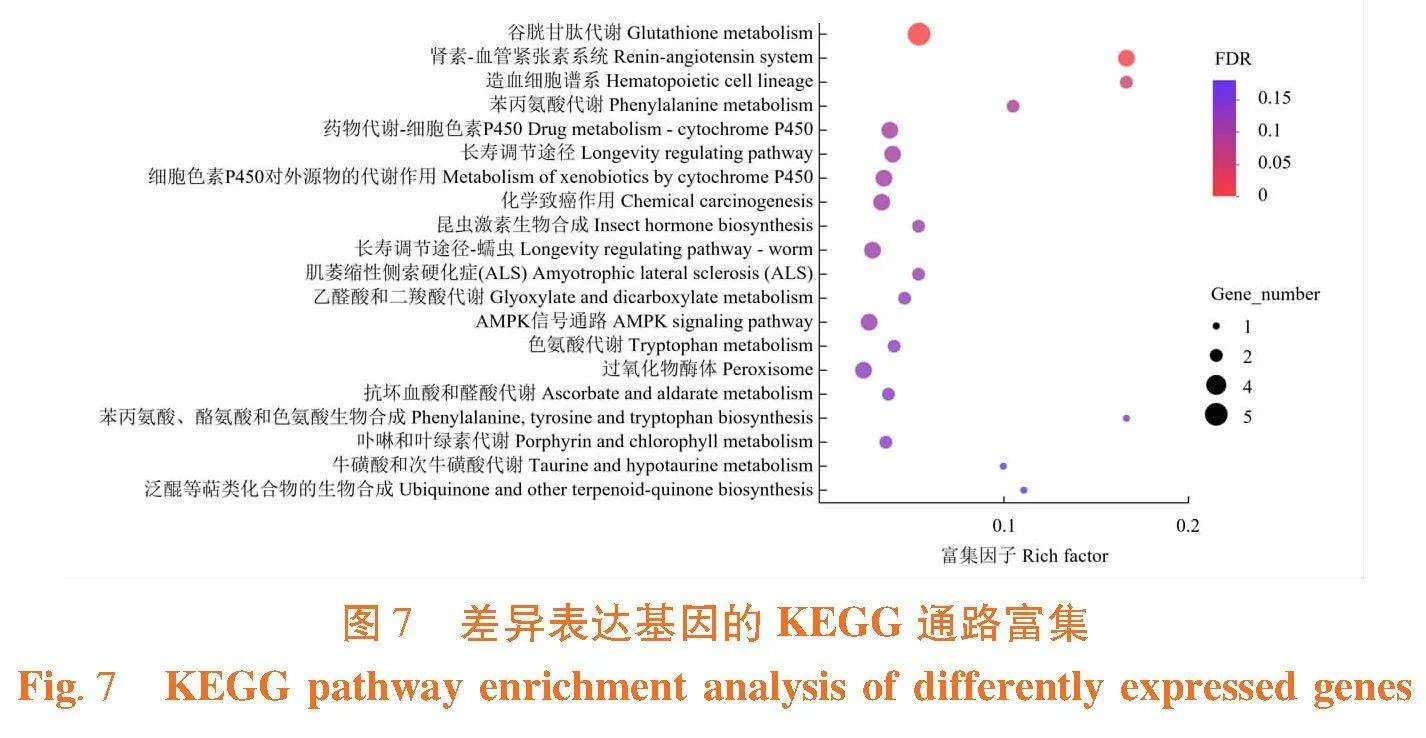
95个差异表达基因主要参与了59个代谢途径,288个上调表达基因中有43个基因(14.90%)参与了29个代谢通路,107个下调表达基因中有52个基因(48.60%)被富集到38个代谢通路中。95个差异表达基因中富集到谷胱甘肽代谢中的基因数目最多,富集也最显著;其次富集显著的是肾素-血管紧张素系统和造血细胞谱系。选择富集最显著的20个KEGG代谢通路在气泡中展示。图7
2.5 气味结合蛋白基因表达水平
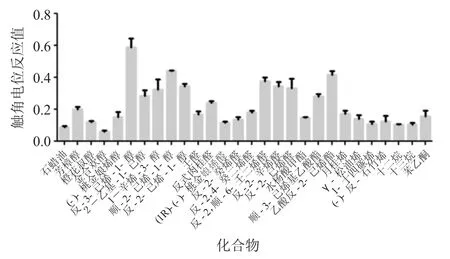
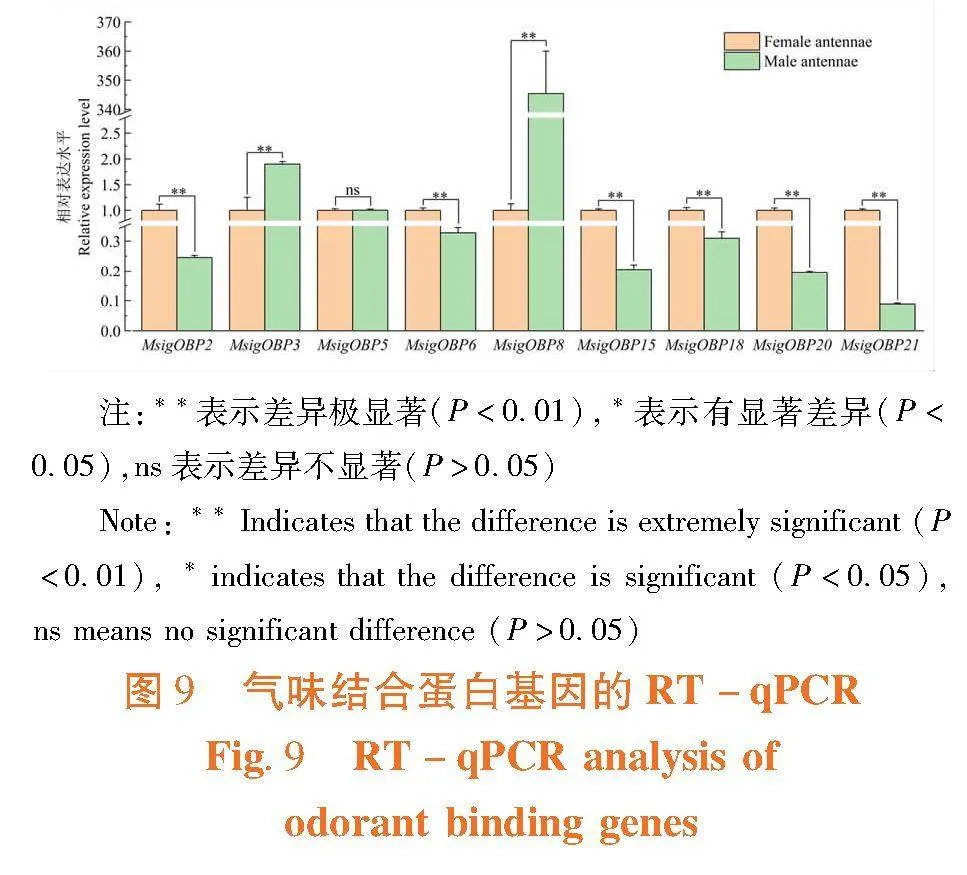
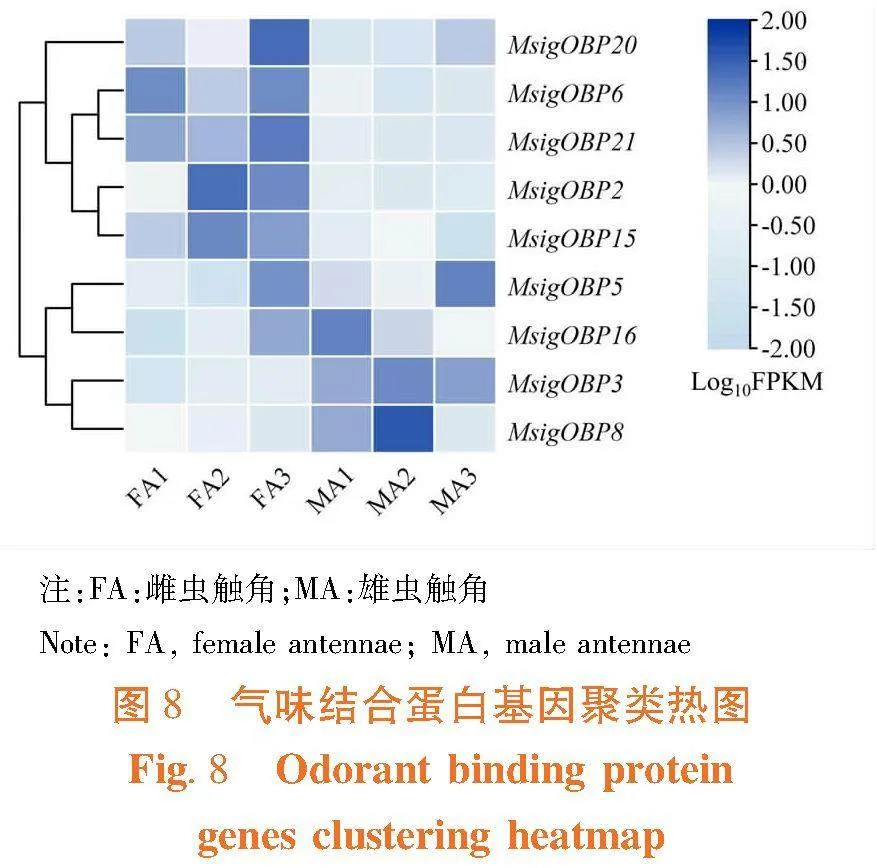
研究表明,OBP基因在双斑长跗萤叶甲雌、雄虫触角中稳定表达,其中MsigOBP2、MsigOBP6、MsigOBP15、MsigOBP18、MsigOBP20和MsigOBP21在双斑长跗萤叶甲雌虫触角中的相对表达水平显著高于雄虫触角,MsigOBP3和MsigOBP6在雄虫触角中的表达水平显著高于雌虫触角,MsigOBP5在雌、雄虫触角中的相对表达水平没有显著差异。OBP基因在雌、雄虫触角中的相对表达水平或变化趋势与转录组测序结果一致,触角是OBP基因发挥作用的主要场所。图9
3 讨 论
3.1
双斑长跗萤叶甲为多食性昆虫,可取食多种农作物[14]。试验利用RT-qPCR技术对筛选出的OBP基因进行了检测,结果表明以上基因均能够在双斑长跗萤叶甲雌、雄虫触角中稳定表达,印证了触角是气味结合蛋白作用的主要场所。
3.2
研究中双斑长跗萤叶甲触角转录组的测序和拼接效果良好,组装优化后得到73 050条unigenes。与和绿豆象Callosobruchus chinensis的触角转录组得到69 847条unigenes处于同一数量水平上[33]。将unigenes与获得的7大数据库比对,有46.86%的unigenes至少在1个数据库中得到注释,基于双斑长跗萤叶甲触角转录组在NR数据库中的注释信息,比对得知匹配双斑长跗萤叶甲unigenes最多的物种是玉米根萤叶甲,玉米根萤叶甲的基因组信息已获得[34]。与多数昆虫转录组测序结果表明注释到的unigenes均与基因组信息已知的亲缘关系较近的物种相似性序列最多相一致,如红脂大小蠹Dendroctonus valens的在NR注释中有69.1%的基因与鞘翅目模式昆虫赤拟谷盗Tribolium castaneum相似性高[35]。
3.3
基于GO数据库功能注释,双斑长跗萤叶甲触角转录组共有19 135条unigenes分别注释于生物过程、分子功能和细胞成分3大类,在这3大类中分别注释到细胞过程、结合和代谢过程的unigenes数量最多,这和七星瓢虫Coccinella septempunctata[36]触角转录组在GO数据库分子功能大类中注释到催化活性的unigenes数量多于结合存在差异,但与花椒窄吉丁Agrilus zanthoxylumi[37]成虫触角在GO数据库注释得到的结果一致,双斑长跗萤叶甲的触角中也存在大量具有与化学信息物质结合的功能蛋白,例如OBPs。在KEGG数据库中注释到信号转导unigenes数量最多,有1146条,占总注释基因的11.4%,触角在双斑长跗萤叶甲在信号转导过程中发挥重要作用。与大多数昆虫相似,双斑长跗萤叶甲触角unigenes在KOG数据库中注释到一般功能预测的数量最多[38-40]。
3.4
某些基因的表达情况在不同性别昆虫或者同性别昆虫的不同组织间存在一定差异,例如鳞翅目中茶谷蛾Agriophara rhombata雌、雄虫触角中筛选得到1 307个差异表达基因,其中474个基因在雄性茶谷蛾触角中上调表达,833个基因在雌性茶谷蛾触角中上调表达[41],大蜡螟Galleria mellonella雌、雄成虫触角中鉴定出了114个差异表达基因,66个基因在雌虫触角中高表达,48个基因在雄虫触角中高表达[42]。鞘翅目中眉斑并脊天牛Glenea cantor Fabricius的雌、雄虫触角转录组中筛选得到了488个差异表达基因,其中268个基因上调表达,220个基因下调表达[43]。双斑长跗萤叶甲雌、雄虫触角中筛选得到395个差异表达基因。以雌虫触角为对照组,在雄虫触角中上调表达的基因有288个,下调表达的基因有107个,少于花椒窄吉丁雌、雄成虫触角中的726个差异表达基因[44],多于不同性别四纹豆象Callosobruchus maculatus (F.)触角中的231个差异表达基因[45]。与花椒窄吉丁相似的是,双斑长跗萤叶甲在雄虫触角中上调表达的基因数量高于雌虫触角,与前期用扫描电镜观察得知雄虫触角上的感器数量明显多于雌虫触角相印证[20]。根据GO功能富集结果可知,双斑长跗萤叶甲雌、雄虫触角的差异表达基因最显著富集的功能为气味结合和嗅觉受体活性,这些差异基因是导致雌虫、雄虫气味感知差异的关键靶标基因。395个差异表达基因只有95个基因在KEGG数据库中获得代谢途径富集,而大部分差异表达基因涉及的代谢通路是未知的,该未知通路的基因有待于进一步研究。
4 结 论
对双斑长跗萤叶甲成虫触角进行了转录组测序并构建了cDNA文库,46.86%的unigenes在至少一个数据库中得到注释信息。在雌虫、雄虫触角中筛选出395个差异表达基因,与雌虫触角相比,288个基因在雄虫触角中上调表达,107个基因下调表达。预测到双斑长跗萤叶甲雌、雄虫中的差异表达基因主要涉及该叶甲的气味识别过程。所选的气味结合蛋白基因可在双斑长跗萤叶甲触角中稳定表达。
参考文献(References)
[1]中国科学院动物研究所昆虫分类区系室叶甲组,河北省张家口地区坝下农业科学研究所植保组,河北省蔚县农业局植保站西合营公社技术站. 双斑萤叶甲研究简报 [J]. 昆虫学报, 1979, 22 (1) : 115-117.Research Group of Leaf Beetle, Division of Insect Taxonomy Institute of Agriculture of Baxia, Changchiakou District, Hopei Province, Technical Station, Plant Protection Station of Xiheying Peoples Commune, Agricultural Bureau of Yu County, Hopei Provinge. A preliminary study of the bionomics of the galerucid beetle, Monolepta hieroglyphica (Motschulsky) [J]. Acta Entomologica Sinica, 1979, 22 (1) : 115-117.
[2]虞佩玉, 王书永, 杨星科. 中国经济昆虫志,第五十四册,鞘翅目,叶甲总科(二)[M]. 北京:科学出版社, 1996.YU Peiyu, WANG Shuyong, YANG Xingke. Economic Insect Fauna of China. Coleoptera: Chrysomeloidea (II) [M]. Beijing: Science Press, 1996.
[3]Wagner T, Bieneck S. Galerucine type material described by victor motschulsky in 1858 and 1866 from the Zoological Museum Moscow (Coleoptera: Chrysomelidae, Galerucinae) [J]. Entomologische Zeitschrift mit Insekten-Brse, 2012, 122 (5) : 205-216.
[4] 梁日霞, 王振营, 何康来, 等. 基于线粒体COⅡ基因序列的双斑长跗萤叶甲中国北方地理种群的遗传多样性研究[J]. 昆虫学报, 2011, 54(7): 828-837.LIANG Rixia, WANG Zhenying, HE Kanglai, et al. Genetic diversity of geographic populations of Monolepta hieroglyphica (Motschulsky) (Coleoptera: Chrysomelidae) from North China estimated by mitochondrial COⅡ gene sequences[J]. Acta Entomologica Sinica, 2011, 54(7): 828-837.
[5] 陈光辉, 尹弯, 李勤, 等. 双斑长跗萤叶甲研究进展[J]. 中国植保导刊, 2016, 36(10): 19-26.CHEN Guanghui, YIN Wan, LI Qin, et al. Research progress on Monolepta hieroglyphica (Motschulsky)[J]. China Plant Protection, 2016, 36(10): 19-26.
[6] 李菁, 张小飞, 徐玲玲, 等. 中国南方双斑长跗萤叶甲地理种群遗传结构及Wolbachia感染[J]. 昆虫学报, 2021, 64(6): 730-742.LI Jing, ZHANG Xiaofei, XU Lingling, et al. Genetic structure and Wolbachia infection in geographical populations of Monolepta hieroglyphica(Coleoptera: Chrysomelidae)in South China[J]. Acta Entomologica Sinica, 2021, 64(6): 730-742.
[7] 赵秀梅, 郑旭, 郭井菲, 等. 齐齐哈尔市玉米田双斑长跗萤叶甲成虫发生规律[J]. 应用昆虫学报, 2021, 58(4): 979-984.ZHAO Xiumei, ZHENG Xu, GUO Jingfei, et al. Occurrence of Monolepta hieroglyphica adults in cornfields in Qiqihar[J]. Chinese Journal of Applied Entomology, 2021, 58(4): 979-984.
[8] 陈静, 张建萍, 张建华, 等. 双斑长跗萤叶甲的嗜食性[J]. 昆虫知识, 2007, 44(3): 357-360.CHEN Jing, ZHANG Jianping, ZHANG Jianhua, et al. Food preference of Monolepta hieroglyphica[J]. Chinese Bulletin of Entomology, 2007, 44(3): 357-360.
[9] He Q, Song X M, Ma H W, et al. The complete mitochondrial genome of Monolepta hieroglyphica (Motschulsky) (Coleoptera: Chrysomelidae)[J]. Mitochondrial DNA Part B, 2021, 6(7): 2019-2021.[LinkOut]
[10] 田永浩, 张建萍, 陈静, 等. 新疆棉花新害虫双斑长跗萤叶甲的发生特点及防治策略[J]. 安徽农学通报, 2007, 13(10): 120-121, 230.TIAN Yonghao, ZHANG Jianping, CHEN Jing, et al. Occurring characteristic and preventions and control strategy of Monolepta hieroglyphica(motschulsky)-a new pest of the cotton field in Xinjiang[J]. Anhui Agricultural Science Bulletin, 2007, 13(10): 120-121, 230.
[11] 杜建军, 云雷. 双斑长跗萤叶甲发生危害特点及防治措施[J]. 陕西农业科学, 2009, 55(3): 202-203.DU Jianjun, YUN Lei. Characteristics and control measures of occurrence and damage of Lepidoptera bimaculata[J]. Shaanxi Journal of Agricultural Sciences, 2009, 55(3): 202-203.
[12] 李广伟, 陈秀琳. 新疆棉区双斑长跗萤叶甲生活习性及消长动态调查研究[J]. 中国植保导刊, 2010, 30(6): 8-10.LI Guangwei, CHEN Xiulin. Studies on biological characteristics and population dynamics of Monolepta hieroglyphica in cotton in Xinjiang[J]. China Plant Protection, 2010, 30(6): 8-10.
[13] Zhang X L, Zhang R Y, Li L, et al. Negligible transcriptome and metabolome alterations in RNAi insecticidal maize against Monolepta hieroglyphica[J]. Plant Cell Reports, 2020, 39(11): 1539-1547.
[14] Zheng F, Jiang H, Jia J L, et al. Effect of dimethoate in controlling Monolepta hieroglyphica (Motschulsky) and its distribution in maize by drip irrigation[J]. Pest Management Science, 2020, 76(4): 1523-1530.
[15] 戴建青, 韩诗畴, 杜家纬. 植物挥发性信息化学物质在昆虫寄主选择行为中的作用[J]. 环境昆虫学报, 2010, 32(3): 407-414.DAI Jianqing, HAN Shichou, DU Jiawei. Progress in studies on behavioural effect of semiochemicals of host plant to insects[J]. Journal of Environmental Entomology, 2010, 32(3): 407-414.
[16]陈秀琳, 李琳琳, 陈玉鑫, 等. 梨小食心虫气味受体GmolOR10基因克隆及表达谱分析 [J/OL]. 植物保护 : 2023, 1-13 [2023-04 -28].CHEN Xiulin, LI Linlin, CHEN Yuxin, et al. Cloning and expression pattern of odorant receptor GmolOR10 in Grapholita molesta Busck [J/OL]. Plant Protection : 2023, 1-13 [2023-04 -28].
[17] Pham H T, McNamara K B, Elgar M A. Age-dependent chemical signalling and its consequences for mate attraction in the gumleaf skeletonizer moth, Uraba lugens[J]. Animal Behaviour, 2021, 173: 207-213.
[18] Segura D F, Belliard S A, Vera M T, et al. Plant chemicals and the sexual behavior of male tephritid fruit flies[J]. Annals of the Entomological Society of America, 2018, 111(5): 239-264.
[19] 赵航, 吴国星, 汤永玉, 等. 叉角厉蝽触角转录组及嗅觉相关基因分析[J]. 环境昆虫学报, 2022, 44(5): 1205-1217.ZHAO Hang, WU Guoxing, TANG Yongyu, et al. Analysis of the antennal transcriptome and olfaction-related genes of Eocanthecona furcellata[J]. Journal of Environmental Entomology, 2022, 44(5): 1205-1217.
[20] 张聪, 王振营, 何康来, 等. 双斑长跗萤叶甲触角感器的扫描电镜观察[J]. 应用昆虫学报, 2012, 49(3): 756-761.ZHANG Cong, WANG Zhenying, HE Kanglai, et al. Scanning electron microscopy studies of antennal sensilla of Monolepta hieroglyphica[J]. Chinese Journal of Applied Entomology, 2012, 49(3): 756-761.
[21] 刘敏, 刘爱萍, 韩海斌. 寄生蜂转录组学研究进展[J]. 植物保护, 2021, 47(2): 11-19.LIU Min, LIU Aiping, HAN Haibin. A review of transcriptome sequencing in parasitoid wasps[J]. Plant Protection, 2021, 47(2): 11-19.
[22] Güell M, van Noort V, Yus E, et al. Transcriptome complexity in a genome-reduced bacterium[J]. Science, 2009, 326(5957): 1268-1271.
[23] 刘伟, 郭光艳, 秘彩莉. 转录组学主要研究技术及其应用概述[J]. 生物学教学, 2019, 44(10): 2-5.LIU Wei, GUO Guangyan, BEI Caili. A summary of the main research techniques of transcriptology and their applications[J]. Biology Teaching, 2019, 44(10): 2-5.
[24] 李资聪, 刘磊, 杨斌, 等. 黄野螟化学感受基因的鉴定与分析[J]. 植物保护, 2021, 47(6): 34-48.LI Zicong, LIU Lei, YANG Bin, et al. Identification and analysis of chemosensory genes in Heortia vitessoides[J]. Plant Protection, 2021, 47(6): 34-48.
[25] 白鹏华, 王冰, 张仙红, 等. 昆虫气味受体的研究方法与进展[J]. 昆虫学报, 2022, 65(3): 1-22.BAI Penghua, WANG Bing, ZHANG Xianhong, et al. Research methods and advances of odorant receptors in insects[J]. Acta Entomologica Sinica, 2022, 65(3): 1-22.
[26] 詹文会, 张苏芳, 耿红卫, 等. 天牛嗅觉感受相关蛋白研究进展[J]. 河南农业科学, 2018, 47(3): 1-6.ZHAN Wenhui, ZHANG Sufang, GENG Hongwei, et al. Research progress on olfactory recognition proteins of longhorn beetle[J]. Journal of Henan Agricultural Sciences, 2018, 47(3): 1-6.
[27] Rondoni G, Roman A, Meslin C, et al. Antennal transcriptome analysis and identification of candidate chemosensory genes of the harlequin ladybird beetle, Harmonia axyridis (Pallas) (Coleoptera: Coccinellidae)[J]. Insects, 2021, 12(3): 209.
[28] 庄翔麟, 吉帅帅, 赵昱杰, 等. 灭字脊虎天牛CSP基因的鉴定、表达谱及XquaCSP7基因的功能研究[J]. 植物保护, 2020, 46(3): 30-39.ZHUANG Xianglin, JI Shuaishuai, ZHAO Yujie, et al. Identification and expression pattern of chemosensory protein genes from Xylotrechus quadripes Chevrolat (Coleoptera: Cerambycidae) and functional studies of XquaCSP7 gene[J]. Plant Protection, 2020, 46(3): 30-39.
[29] Wang X, Wang S, Yi J K, et al. Three host plant volatiles, hexanal, lauric acid, and tetradecane, are detected by an antenna-biased expressed odorant receptor 27 in the dark black chafer Holotrichia parallela[J]. Journal of Agricultural and Food Chemistry, 2020, 68(28): 7316-7323.
[30] Yuvaraj J K, Roberts R E, Sonntag Y, et al. Putative ligand binding sites of two functionally characterized bark beetle odorant receptors[J]. BMC Biology, 2021, 19(1): 16.
[31] Hong B, Chang Q, Zhai Y J, et al. Functional characterization of odorant binding protein PyasOBP2 from the jujube bud weevil, Pachyrhinus yasumatsui (Coleoptera: Curculionidae) 2022.
[32] Grabherr M G, Haas B J, Yassour M, et al. Full-length transcriptome assembly from RNA-Seq data without a reference genome[J]. Nature Biotechnology, 2011, 29(7): 644-652.
[33]Zhang Y N, Kang K, Xu L, et al. Deep sequencing of antennal transcriptome from Callosobruchus chinensis to characterize odorant binding protein and chemosensory protein genes [J]. Journal of Stored Products Research, 2017, 74 : 13-21.
[34] 黄聪, 李有志, 杨念婉, 等. 入侵昆虫基因组研究进展[J]. 植物保护, 2019, 45(5): 112-120, 134.HUANG Cong, LI Youzhi, YANG Nianwan, et al. Progresses in invasive insect genomics[J]. Plant Protection, 2019, 45(5): 112-120, 134.
[35] Gu X C, Zhang Y N, Kang K, et al. Antennal transcriptome analysis of odorant reception genes in the red turpentine beetle (RTB), Dendroctonus valens[J]. PLoS One, 2015, 10(5): e0125159.[PubMed]
[36] 杨雪娇, 张蔓, 江宇航, 等. 七星瓢虫触角转录组及嗅觉相关基因分析[J]. 昆虫学报, 2020, 63(6): 717-726.YANG Xuejiao, ZHANG Man, JIANG Yuhang, et al. Analysis of the antennal transcriptome and olfaction-related genes of Coccinella septempunctata (Coleoptera: Coccinellidae)[J]. Acta Entomologica Sinica, 2020, 63(6): 717-726.
[37] 巩雪芳, 谢寿安, 杨平, 等. 花椒窄吉丁触角转录组及嗅觉相关基因分析[J]. 昆虫学报, 2020, 63(10): 1159-1170.GONG Xuefang, XIE Shouan, YANG Ping, et al. Analysis of the antennal transcriptome and olfaction-related genes of Agrilus zanthoxylumi (Coleoptera: Buprestidae)[J]. Acta Entomologica Sinica, 2020, 63(10): 1159-1170.
[38] 胡佳萌, 徐丹萍, 卓志航, 等. 云斑天牛成虫触角转录组及嗅觉相关基因分析[J]. 应用昆虫学报, 2019, 56(5): 1037-1047.HU Jiameng, XU Danping, ZHUO Zhihang, et al. Analysis of antennal transcriptome and olfaction-related genes of adult Batocera horsfieldi (Hope)[J]. Chinese Journal of Applied Entomology, 2019, 56(5): 1037-1047.
[39] 宋月芹, 董钧锋, 陈庆霄, 等. 点蜂缘蝽触角转录组及化学感受相关基因的分析[J]. 昆虫学报, 2017, 60(10): 1120-1128.SONG Yueqin, DONG Junfeng, CHEN Qingxiao, et al. Analysis of the antennal transcriptome and chemoreception-related genes of the bean bug, Riptortus pedestris (Hemiptera: Alydidae)[J]. Acta Entomologica Sinica, 2017, 60(10): 1120-1128.
[40] 张健, 谢婉莹, 董曼羽, 等. 青杨天牛嗅觉相关基因的鉴定及表达谱分析[J]. 江苏农业科学, 2022, 50(4): 23-28.ZHANG Jian, XIE Wanying, DONG Manyu, et al. Identification and expression profile analysis of olfactory related genes of Anoplophora populnea[J]. Jiangsu Agricultural Sciences, 2022, 50(4): 23-28.
[41] 龙亚芹, 罗梓文, 王雪松, 等. 茶谷蛾成虫触角转录组及嗅觉相关基因分析[J]. 茶叶科学, 2021, 41(4): 553-563.LONG Yaqin, LUO Ziwen, WANG Xuesong, et al. Analysis of the antennal transcriptome and olfactory-related genes in the Agriophara rhombata[J]. Journal of Tea Science, 2021, 41(4): 553-563.
[42] 杨爽, 赵慧婷, 徐凯, 等. 大蜡螟触角转录组及嗅觉相关基因分析[J]. 应用昆虫学报, 2019, 56(6): 1279-1291.YANG Shuang, ZHAO Huiting, XU Kai, et al. Analysis of the antennal transcriptome and olfaction-related genes of the greater wax moth Galleria mellonella(Lepidoptera: Pyralidae)[J]. Chinese Journal of Applied Entomology, 2019, 56(6): 1279-1291.
[43] Wu G X, Su R R, Ouyang H L, et al. Antennal transcriptome analysis and identification of olfactory genes in Glenea cantor Fabricius (Cerambycidae: Lamiinae)[J]. Insects, 2022, 13(6): 553.
[44] 巩雪芳, 谢寿安, 杨平, 等. 花椒窄吉丁雌、雄成虫触角差异表达基因的初步筛选[J]. 农业生物技术学报, 2019, 27(7): 1233-1245.GONG Xuefang, XIE Shouan, YANG Ping, et al. Preliminary screening of differentially expressed genes of female and male antenna of Agrilus zanthoxylumi[J]. Journal of Agricultural Biotechnology, 2019, 27(7): 1233-1245.
[45] Tanaka K, Shimomura K, Hosoi A, et al. Antennal transcriptome analysis of chemosensory genes in the cowpea beetle, Callosobruchus maculatus (F.)[J]. PLoS One, 2022, 17(1): e0262817.