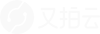



摘要: "ICE1是bHLH类转录因子,能够特异性结合CBF3基因启动子序列中的元件,从而激活下游冷诱导基因的表达,在响应低温胁迫时具有重要作用。本文从低温条件下大青杨(Populus ussuriensis)cDNA中克隆得到一个受低温诱导的PuICE1基因,利用生物信息学工具分析PuICE1的序列和编码蛋白特征、利用实时荧光定量PCR(qRT-PCR)分析PuICE1基因在低温胁迫条件下的表达模式。结果表明:大青杨PuICE1基因的cDNA全长序列长1 635 bp,含有完整的开放阅读框,可编码545个氨基酸。PuICE1编码的蛋白序列含有1个典型的HLH结构域,属于bHLH类转录因子,进化树构建结果表明,大青杨PuICE1与毛果杨(P.trichocarpa)和甜杨(P. suaveolens)ICE1存在着较高的同源性。通过qRT-PCR分析表明,在4 ℃低温胁迫条件下,随着胁迫处理时间(0、1.5、3、6、12、24、48、72 h)的延长,大青杨根、茎、叶片中的PuICE1基因表达量都是在72 h时达到峰值,而在芽中PuICE1基因表达量呈现先下降后上升的趋势,在48 h表达量达到峰值。
关键词: "大青杨; "PuICE1; "生物信息学分析
中图分类号: " S 792.113 " " " " " " " 文献标识码: " A " " " " " " " "文章编号:1001 - 9499(2024)04 - 0023 - 06
Cloning and Bioinformatics Analysis of PuICE1 in Populus ussuriensis
WANG Yanmin1 , 2 , 3 LI Jing2 , 3 YIN Zhihui2 , 3 LI Zhenghua2 , 3
GUO Chengbo2 , 3 LI Haixia2 , 3 BAI Hui2 , 3**
(1. "Forestry Research Academy of Heilongjiang Province, "Heilongjiang Harbin 150081; "2. "Forestry Research Institute of Heilongjiang Province, "Heilongjiang Harbin 150081; "3. "Key Laboratory of Fast-Growing Tree Cultivating of Heilongjiang Province, "Heilongjiang Harbin 150081)
Abstract ICE1 encodes a MYC-like bHLH transcriptional activator, which could specially band to active domain of CBF3 promoter and induce transcriptal expression of cold-responsive genes downstream of CBF3,play important roles in cold stress response. We cloned ICE1 in Populus ussuriensis using cDNA after 4 ℃ low temperature treatment of 24 hours, and named PuICE1.A 1 635 bp cDNA clone was obtained in our study, which contains a putatively entire open reading frame (ORF) and encodes a MYC-like protein of 545 amino acids. Bioinformatic analysis revealed that this cDNA was highly homologous to ICE1 from P.trichocarpa and P.suaveolens, indicating that the interest cDNA was PuICE1 gene. The result of qRT-PCR analysis showed that with the extension of treatment time(0, 1.5, 3, 6, 12, 24, 48, 72 h) at 4 ℃, the gene expression in roots, stems and leaves reached its peak at 72h, while the gene expression in buds showed a trend of first decreasing and then increasing, and reached its peak at 48 h.
Key words Populus ussuriensis; PuICE1; bioinformatics analysis
大青杨(Populus ussuriensis)属落叶乔木[ 1 ],主要产地位于35°~55°N左右,海拔300~1 400 m,喜光、偏湿,大青杨木质白且密,软而轻,耐腐朽,速生、优质、适应性强,易于繁殖、用途广[ 2 ]。杨树作为重要的木本植物之一,在生产发育阶段也面临着各种生物或非生物胁迫的影响。干旱、高温、低温、损伤等非生物胁迫严重影响植物的生长发育及产量。低温胁迫严重限制了植物的生长发育和地理分布,影响作物的产量和质量[ 3 - 6 ]。植物在漫长的演变过程中,已经进化出精确的细胞和分子机制来提高它们在极端低温条件下的生存机会[ 4 , 6 - 8 ]。了解植物如何通过感知、转导和响应冷信号来适应冷胁迫,对于培育抗低温作物至关重要[ 4 , 9 ]。
植物跨膜蛋白COLD1被鉴定为一个感知冷信号的传感器,它与G-protein α subunit1 (RGA1)形成复合物,介导细胞内Ca2+流动,以提高cold-
regulated(COR)基因的表达[ 4, 10 ]。COR基因编码的蛋白可以通过稳定膜结构来抵抗冻害[ 11 , "12 ]。C-repeat binding factors (CBFs)是能够与COR基因启动子中的CRT/DRE元件结合从而诱导其表达的关键转录因子(TFs),CBFs转录因子本身在低温非冰冻条件下会迅速上调[ 11 , 13 ]。在过去的20多年里,多种TFs被证明可以调节CBF的表达[ 4 ],其中,inducer of CBF expression 1 (ICE1)是能够诱导CBF基因表达的关键转录因子之一[ 4, 14 - 16 ]。ICE1是一类MYC-
like basic helix-loop-helix (bHLH) 转录因子,在冷胁迫条件下直接与CBF基因启动子中的MYC序列(CANNTG)结合[ 14 , 15 , 17 , 18 ]。与CBFs和CORs相比,ICE1是一个组成型表达基因,在转录水平上对冷胁迫没有明显的响应[ 4 , "14 ]。然而,泛素化、聚合化和磷酸化等多种翻译后修饰对低温条件下ICE1的转录调控起着重要作用[ 19 - 23 ]。
本研究利用RT-PCR方法从大青杨中克隆PuICE1基因,对大青杨PuICE1基因及其蛋白质进行生物信息学分析,并研究该基因在低温胁迫下的表达模式,从而为研究大青杨PuICE1基因的功能研究提供基础和线索。
1 试验材料及方法
1. 1 试验材料
对大青杨组培苗进行4 ℃低温处理24 h。
胶回收试剂盒购于Omega公司;反转录试剂盒、Ex Taq 聚合酶、dNTP、pGEM-T Easy载体、T4连接酶购于TaKaRa公司;大肠杆菌菌株DH5α购自天根生物技术公司。所用引物及测序委托上海生工生物工程技术服务有限公司(Sangon,http://www.sangon.biogo.net/)合成及完成。
1. 2 试验方法
1. 2. 1 大青杨总RNA提取
采用百泰克公司通用植物总RNA大量提取试剂盒(RP3312)提取大青杨总RNA,反转录cDNA用于RT-PCR。
1. 2. 2 大青杨PuICE1基因克隆
根据毛果杨(P.trichocarpa)基因组(http://www.phytozome.net/poplar)中PtICE1基因序列设计特异引物(表1),以4 ℃低温处理大青杨cDNA为模板,进行RT-PCR。PCR扩增体系:cDNA2.0 μL,10×Ex Taq PCR buffer2.0 μL,10 mmol/L dNTP Mix0.4 μL,10 μmol/L引物各1.0 μL,5 U/μL Ex Taq0.2 μL,补水至20.0 μL。PCR反应程序:94 ℃预变性30 s;94 ℃变性10 s、58 ℃退火30 s、72 ℃延伸2 min,共35个循环;72 ℃延伸10 min,4 ℃保存。取5 μ
LPCR产物进行电泳检测,以DL2000 DNA Marker为参照。回收PCR产物,克隆至pGEM-T Easy载体。挑取阳性克隆送至上海生工生物工程技术服务有限公司测序。
1. 2. 3 大青杨PuICE1序列分析
运用NCBI网站中的ORF Finder程序(http://www.ncbi.nlm.nih.gov/gorf/gorf.html)查找PuICE1基因的开放阅读框。ProtParam程序(http://au.expasy.org/tools/protparam.html)用于蛋白分子量、理论等电点、半衰期、脂肪系数、不稳定系数、总平均疏水性的预测。利用Expasy工具(http://au.expasy.org/tools)中提供的ProtScale软件在线分析氨基酸的亲、疏水性,通过Pfam27.0(http://pfam.sanger.ac.uk/)和protein blast (http://blast.ncbi.nlm.nih.gov/Blast.cgi)查找其保守结构域,选取10个其他物种的ICE1蛋白质用Bioedit软件进行多序列比对,并用MEGA软件构建系统发育树。
1. 2. 4 大青杨PuICE1基因表达模式分析
提取低温胁迫下大青杨不同组织部位(芽、叶、茎、根)RNA,用反转录试剂盒逆转录为cDNA作为qRT-PCR模板,检测低温胁迫条件下大青杨不同组织部位在不同胁迫时间下的表达情况。根据克隆得到的PuICE1基因的ORF序列,使用Primer Premier5.0软件设计定量分析引物PuICE1-RT-F和PuICE1-RT-R(表1),以大青杨PuActin基因作为内参基因,其引物为PuActin-F和PuActin-R(表1),进行实时荧光定量PCR(quantitative real-time PCR, qRT-PCR)分析。PCR反应体系为:SYBR Premix Ex Taq
TMⅡ10μL,正、反向引物各0.5 μL,ROX Reference " Dye(50×)0.4 μL,cDNA 模板2.0 μL,ddH2O6.6 μL。PCR扩增程序为:95 ℃预变性30 s;95 ℃变性5 s,60 ℃退火30 s,共40个循环。试验设3次重复,采用2-ΔΔCt法计算基因的相对表达量。
2 结果与分析
2. 1 PuICE1基因序列结构分析
以4 ℃低温处理24 h的cDNA为模板,利用设计的ICE-F和ICE-R引物,进行PCR扩增,得到一条1.6 kb左右的特异片段(图1),胶回收特异片段,连接T载体,进行大肠杆菌转化,挑取阳性克隆,对阳性克隆进行PCR验证,送样测序,对测序结果进行分析。结果显示,该基因开放读码框1 635 bp,推测其编码一个含545个氨基酸,相对分子量为134 704.2,理论等电点(pI)为5.00、脂肪系数为28.93、不稳定系数为39.63,是稳定蛋白(参数大于40是不稳定蛋白)。利用Expasy工具(http://au.expasy.org/tools)中提供的ProtScale软件在线分析氨基酸的亲、疏水性,氨基酸残基总平均疏水性(GRAVY)为0.701,说明蛋白疏水性较强。对其蛋白序列进行比对分析(http://pfam.sanger.ac.uk/search/sequence),发现在序列C端1084-1218碱基序列之间存在一个HLH(Helix-loop-helix DNA-binding domain)结构域(图2)。
2. 2 PuICE1蛋白序列分析
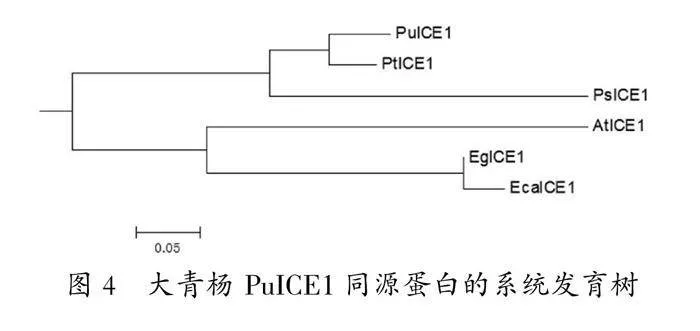
利用Blast程序(http://www.ncbi.nlm.nih.gov/BLAST/)进行序列同源性搜索,选择与其相似程度高的其他6种不同植物的ICE1氨基酸序列,用多序列联配程序Clustalx进行多序列比对。所选择的5种植物的ICE1基因分别为:拟南芥(Arabidopsis thaliana)、毛果杨、甜杨(P. suaveolens)、蓝桉树(Eucalyptus globulus)、赤桉(Eucalyptus camaldulensis)。通过Clustal软件对该基因编码蛋白序列与其他相似程度高的5种植物的ICE1序列同源性比较发现,大青杨PuICE1蛋白序列与其他5种植物的ICE1蛋白的同源性分别为49%、87%、71%、55%、54%,其中与毛果杨的同源性最高。用Bioedit软件对上述ICE1序列做多序列比对分析(图4),可以看到ICE1保守结构域序列与其他物种同类的ICE1蛋白有着非常高的序列一致性。多重比对结果显示,大青杨ICE1蛋白与其他植物ICE1蛋白在N端差异较大,但它们在bHLH功能域和C端高度保守。
用MEGA6软件构建了不同植物ICE1的系统发育树(图3),结果显示,ICE1基因家族按照其保守结构的不同发现大青杨的ICE1基因与毛果杨关系较近,而与拟南芥、蓝桉树、赤桉亲缘关系较远。
2. 3 大青杨PuICE1基因的表达模式分析
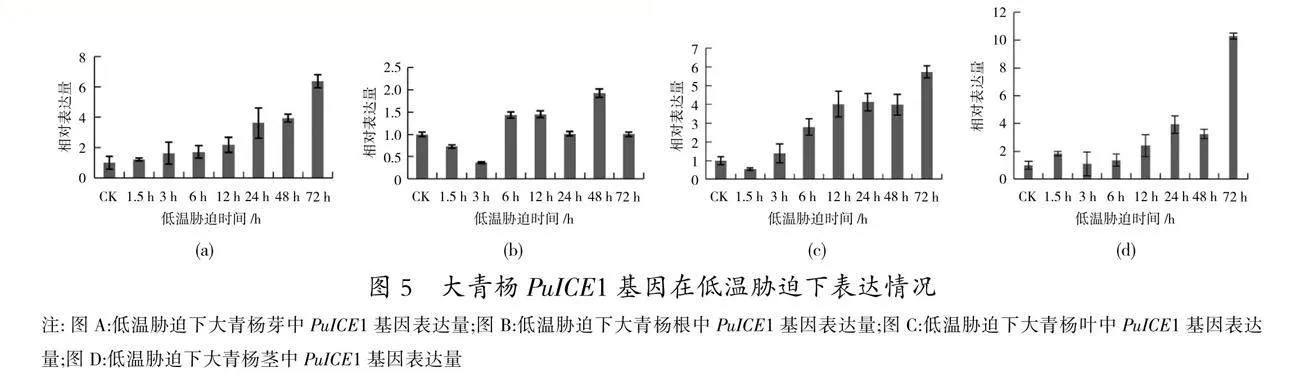
对移栽1个月左右的野生型大青杨进行4 ℃低温胁迫,胁迫时间分别为0、1.5、3、6、12、24、48、72 h,分析在这几个不同胁迫时间下的PuICE1基因表达模式(图5)。4 ℃低温胁迫下,PuICE1基因在大青杨芽中表达量处于先降后升的趋势,在胁迫48 h时的表达量最高,随后表达量有所下降。PuICE1基因在大青杨叶片、茎段、根中的表达量逐渐升高,胁迫72 h时的表达量最高,整体上每个时间点的表达量处于稳定上升的趋势。
3 讨 论
ICE1(inducer of CBF expression1)也被称为诱导CBF表达基因,是能够调节冷诱导基因(cold- regulated,COR)表达的最上游转录因子。它编码一个类似转录激活基因(MYC)的bHLH转录因子,在常温下钝化,在低温情况下能够特异地结合CBF3启动子中的MYC作用元件,诱导CBF3的表达,CBF3进一步结合到其下游目的基因启动子的DRE序列,诱导下游一系列冷诱导基因以及其他在植物寒冷适应中起作用的基因的表达,继而提高转基因植株的抗寒性[ 14 , 24 ]。
本研究根据毛果杨PtICE1基因序列设计特异引物克隆大青杨PuICE1基因,对其进行生物信息学分析,序列比对结果显示,大青杨PuICE1基因与其他物种的ICE1转录因子相似度较高,其中毛果杨87%、甜杨71%,用Bioedit软件对上述ICE1序列做多序列比对分析,可以看到ICE1保守结构域序列与其他物种同类的ICE1蛋白序列一致性较高。多重比对结果表明,大青杨ICE1蛋白与其他植物ICE1蛋白相比,在N端存在较大差异,但它们在bHLH功能域以及C端都具有高度保守的特性。此外,在一个由11个氨基酸组成的区域中,大青杨ICE1与其他物种相比(除甜杨ICE1外)均完全一致,另外,大青杨ICE1中也存在SUMO结合位点,且与毛果杨ICE1完全一致,与甜杨的ICE1同源序列有较小差异,但与拟南芥等的ICE1差异明显,这说明大青杨的在苏素化作用途径中大青杨ICE1作用原理与毛果杨和甜杨类似,而有别于拟南芥等植物。进一步用MEGA软件构建了不同植物PuICE1基因的系统发育树,结果显示,ICE1基因家族按照其保守结构的不同发现大青杨的PuICE1基因与毛果杨亲缘关系较近。
分离并克隆基因是研究基因功能及其表达调控机理的前提基础,与传统生物学研究相比,利用生物信息学方法对基因进行比较研究,可在较快时间获得可靠结果。本研究从大青杨中克隆PuICE1基因,对该基因和其蛋白进行生物信息学分析,从而为大青杨PuICE1基因和蛋白功能研究提供重要依据。
参考文献
[1] 刘晓, " 杨佳, " 张馨, " 等. "大青杨PubZIP1基因的克隆及亚细胞定位与抗旱表达特性分析 [J]. 植物研究, 2020, 40(02): 233 - 242.
[2] 谢政文, "王连军, "陈锦洋, "等. "植物WRKY转录因子及其生物学功能研究进展[J]. 中国农业科技导报, 2016, 18(3): 46 - 54.
[3] Krasensky J, Jonak C. Drought, salt, and temperature stress-induced metabolic rearrangements and regulatory networks[J]. Journal of Experimental Botany, 2012, 63(4): 1593 - 1608.
[4] Shi Y, Ding Y, Yang S. Molecular Regulation of CBF Signaling in Cold Acclimation[J]. Trends in Plant Science, 2018, 23(7): 623 - 637.
[5] Ding Y, Shi Y, Yang S. Molecular Regulation of Plant Responses to Environmental Temperatures [J]. Molecular Plant, 2020, 13(4): 544 - 564.
[6] Ding Y, Yang S. Surviving and thriving: How plants perceive and respond to temperature stress[J]. Developmental Cell, 2022, 57(8): 947 - 958.
[7] 朱健康, "倪建平. "植物非生物胁迫信号转导及应答[J]. 中国稻米, 2016, 22(6): 52 - 60.
[8] 蒋悦, "罗倩, "姜超英, "等. "烟草转录因子NtMYB4a响应干旱、低温和茉莉酸甲酯胁迫的功能分析[J]. 作物研究, 2021, 35(1): 43 - 49.
[9] Chen X, Ding Y, Yang Y, et al. Protein kinases in plant responses to drought, salt, and cold stress[J]. Journal of Integrative Plant Biology,
2021, 63(1): 53 - 78.
[10] Ma Y, Dai X, Xu Y, et al. COLD1 confers chilling tolerance in rice[J]. Cell, 2015, 160(6): 1209 - 1221.
[11] Jaglo-Ottosen K R, Gilmour S J, Zarka D G, et al. Arabidopsis CBF1 overexpression induces COR genes and enhances freezing tolerance[J]. Science, 1998, 280(5360): 104 - 106.
[12] Thomashow M F. PLANT COLD ACCLIMATION: Freezing
Tolerance Genes and Regulatory Mechanisms[J]. Annual Review of Plant Physiology and Plant Molecular Biology, 1999, 50: 571 - 599.
[13] Song Y, Zhang X, Li M, et al. The direct targets of CBFs: In cold stress response and beyond[J]. Journal of Integrative Plant Biology, 2021, 63(11): 1874 - 1887.
[14] Chinnusamy V, Ohta M, Kanrar S, et al. ICE1: a regulator of cold- "induced transcriptome and freezing tolerance in Arabidopsis[J]. Genes Development, 2003, 17(8): 1043 - 1054.
[15] Kim Y S, Lee M, Lee J H, et al. The unified ICE-CBF pathway provides a transcriptional feedback control of freezing tolerance during cold acclimation in Arabidopsis[J]. Plant Molecular Biology, 2015, 89(1 - 2): 187 - 201.
[16] Ding Y, Shi Y, Yang S. Advances and challenges in uncovering cold tolerance regulatory mechanisms in plants[J]. New Phytologist, 2019, 222(4): 1690 - 1704.
[17] Maruyama K, Todaka D, Mizoi J, et al. Identification of cis-acting promoter elements in cold- and dehydration-induced transcrip- tional pathways in Arabidopsis, rice, and soybean[J]. DNA " Research, 2012, 19(1): 37 - 49.
[18] Tang K, Zhao L, Ren Y, et al. The transcription factor ICE1 functions in cold stress response by binding to the promoters of CBF and COR genes[J]. Journal of Integrative Plant Biology, 2020, 62(3): 258 - 263.
[19] Dong C H, Agarwal M, Zhang Y, et al. The negative regulator of plant cold responses, HOS1, is a RING E3 ligase that mediates the ubiquitination and degradation of ICE1[J]. Proceedings of the National Academy of Sciences of The United States of America, 2006, 103(21): 8281 - 8286.
[20] Miura K, Jin J B, Lee J, et al. SIZ1-mediated sumoylation of ICE1 controls CBF3/DREB1A expression and freezing tolerance in Arabidopsis[J]. Plant Cell, 2007, 19(4): 1403 - 1414.
[21] Ding Y, Li H, Zhang X, et al. OST1 kinase modulates freezing tolerance by enhancing ICE1 stability in Arabidopsis[J]. Develo-
pmental Cell, 2015, 32(3): 278 - 289.
[22] Li H, Ding Y, Shi Y, et al. MPK3-and MPK6-Mediated ICE1 Phosphorylation Negatively Regulates ICE1 Stability and Freezing Tolerance in Arabidopsis[J]. Developmental Cell, 2017, 43(5): 630-42.e4.
[23] Ye K, Li H, Ding Y, et al. BRASSINOSTEROID-INSENSITIVE2 Negatively Regulates the Stability of Transcription Factor ICE1 in Response to Cold Stress in Arabidopsis[J]. Plant Cell, 2019, 31 " "(11): 2682 - 2696.
[24] Badawi M, Reddy Y V, Agharbaoui Z, et al. Structure and functionalanalysis of wheat ICE (inducer of CBF expression) genes[J]. Plant Cell Physiol, 2008, 49(8): 1237 - 1249.