摘 要:【目的】研究4-香豆酸辅酶A连接酶(4CL)家族基因Gh4CL30的生物学功能,为棉花株型育种提供理论依据和基因种质资源。
【方法】利用病毒诱导的基因沉默技术和基因编辑技术获得Gh4CL30沉默和编辑植株,测定该基因抑制及敲除植株的黄酮和木质素含量,调查棉花田间表型性状、种子大小和纤维品质。
【结果】Gh4CL30基因沉默和敲除植株中木质素合成相关基因表达量显著下降,4-香豆酸辅酶A连接酶含量显著减少,茎秆中木质素含量显著降低,其株高、种子大小和纤维长度显著降低。
【结论】Gh4CL30通过调控棉花木质素生物合成功能影响其生长发育。
关键词:陆地棉;4-香豆酸辅酶A连接酶;木质素;基因编辑;株高
中图分类号:S188"" 文献标志码:A"" 文章编号:1001-4330(2024)06-1301-09
0 引 言
【研究意义】株高影响棉花的结铃数和纤维品质,适当的株高对棉花机械化采摘尤为重要,因此塑造理想的棉花株型,有利于机采棉高产稳产。目前对棉花株高的研究主要集中于外施激素[1-3],其分子调控网络仍有待进一步探索,挖掘调控株高的关键基因,对培育理想株型的棉花品种具有重要意义。
【前人研究进展】苯丙烷代谢途径是一种植物特异性代谢途径,可将芳香族氨基酸苯丙氨酸或酪氨酸转化为多种次级代谢产物,包括木质素、香豆素和类黄酮等[4,5],在调节植物生长发育、响应胁迫等方面发挥重要作用[6-8]。绿豆中抑制苯丙烷途径中关键酶的合成会导致绿豆幼苗的木质素含量下降,木质部导管塌陷,生长受阻[9,10]。作为苯丙烷途径中最后一个关键的限速酶,4-香豆酸辅酶A连接酶(4CL)介导羟基肉桂酸的激活,在木质素生物合成途径中起着关键作用[11]。杨树幼苗中4CL基因的表达,导致其木质素含量下降,而且茎高、节间长度、直径和体积均显著降低[12]。在烟草中过表达水曲柳Fm4CL基因,其木质素含量显著增加,木质部细胞壁的厚度明显提高[13]。4CL基因家族已在拟南芥[14]、水稻[15]和白杨[16]等植物中被鉴定,根据基因结构和功能的差异被分为I型和II型[17]。I型4CL基因主要参与木质素生物合成,而II型基因则主要参与除了木质素以外的苯丙烷类代谢产物的生物合成。在拟南芥中,At4CL1、At4CL2和At4CL4属于I型,At4CL3属于II型[17]。丹参中I型的Sm4CL1和Sm4CL2主要参与木质素生物合成,II型的Sm4CL3参与类黄酮生物合成[18]。在棉花中4CLs的研究主要集中于耐旱性和抗病性,Gh4CL7沉默显著降低棉花耐旱性,在拟南芥中过表达能显著提高其耐旱性[19]。
【本研究切入点】4-香豆酸辅酶A连接酶(4CL)在木质素生物合成途径中起着关键作用[11]。但4CLs在调控棉花株高中的功能尚不清楚。目前已在陆地棉中鉴定出34个Gh4CL基因[19],试验研究获得I型4CL基因Gh4CL30(GH_D10G0525),需分析其基因表达模式,构建该基因的VIGS沉默和基因敲除载体,通过侵染棉花幼苗和遗传转化,获得基因沉默和敲除突变体植株,分析其在调控棉花木质素合成以及影响株高中的功能。【拟解决的关键问题】利用病毒诱导的基因沉默技术和基因编辑技术获得Gh4CL30沉默和编辑植株,解析Gh4CL30基因在棉花株高发育中的作用,为棉花株型育种提供理论依据和基因资源。
1 材料与方法
1.1 材 料
陆地棉(Gossypium hirsutum L.)品种新彩棉7号由石河子大学农学院棉花遗传育种课题组提供。棉花种植于人工气候室,设定培养条件:温度28℃/23℃(光照/黑暗),湿度为60%,光周期16 h光照/8 h黑暗。田间试验在石河子大学棉花研究所试验地进行。
1.2 方 法
1.2.1 Ⅰ型Gh4CL家族基因表达模式
陆地棉的注释蛋白序列[20]从CottonGen(https://www.cottongen.org/)下载。陆地棉中Gh4CL基因各组织部位的表达量数据从COTTONOMICS(http://cotton.zju.edu.cn/index.htm)下载。Gh4CL基因分类和命名参考文献[19]。TBtools[21]的HeatMap函数用于分析基因表达数据。
1.2.2 基于病毒诱导基因沉默(VIGS)载体构建及棉花侵染
烟草花叶病毒(TRV)载体用于VIGS试验。TRV:GhCHLI作为阳性对照。TRV空载体(TRV:00)作为阴性对照。利用PCR扩增技术,以陆地棉的茎秆cDNA为模板,扩增出Gh4CL30基因约400bp的目的片段,用于构建TRV:Gh4CL30载体。VIGS试验方案参考文献[22]。当TRV:GhCHLI侵染的棉花叶片出现白化现象时,从棉花茎中提取RNA以测定目的基因沉默效率。
1.2.3 基于CRISPR/Cas9基因编辑载体构建与棉花转化
运用CRISPR/Cas9基因编辑系统,载体为pRGEB32-GhU6.9-NPT II(实验室保存)。在Gh4CL30编码区设计2个靶向sgRNA,通过重叠延伸PCR将其整合到单个融合片段中。使用ClonExpress II一步克隆试剂盒(Vazyme,中国南京)将融合片段连接到Bsa I酶切的pRGEB32-GhU6.9-NPT II载体中。将构建的载体pCRISPR-Gh4CL30通过电击转化到根癌农杆菌LBA4404菌株中,用于棉花转化。以新彩棉7号作为Gh4CL30基因编辑的转化受体,将棉种剥壳后用1‰升汞消毒清洗放置在MS培养基上,在28℃暗环境下培养6 d形成无菌幼苗,取幼苗的下胚轴切成1 cm左右小段,菌液(OD值在0.5~0.8)侵染10 min,将下胚轴小段在培养基中培养,形成胚性愈伤组织后放入分化培养基中,直到养成幼苗。PCR检测阳性植株,通过Gh4CL30特异性靶位点测序筛选突变体。
1.2.4 棉花茎秆的细胞学观察及黄酮和木质素含量测定
收集野生型及转基因棉花茎秆样品在液氮中快速粉碎,使用木质素含量测试盒(comin,MZS-2-G)和植物黄酮含量测试盒(mlbio-Good-elisakit)测定木质素和类黄酮含量。从材料的相同节间截取茎段,切成薄片,并在间苯三酚溶液(5g间苯三酚溶于100 mL 95%乙醇中)中浸泡5 min,在18%盐酸中浸泡5 min,随后固定在载玻片上,并立即在显微镜(SteREO Discovery.V20;蔡司)下观察拍照,红色表示木质素的存在。将样品固定在FAA(5%福尔马林、5%冰醋酸、70%酒精)中,制备成石蜡切片,并用蔡司Axio Scope A1显微镜拍摄,观察其细胞结构[23]。
2 结果与分析
2.1 Ⅰ型Gh4CL基因生物信息学及表达模式
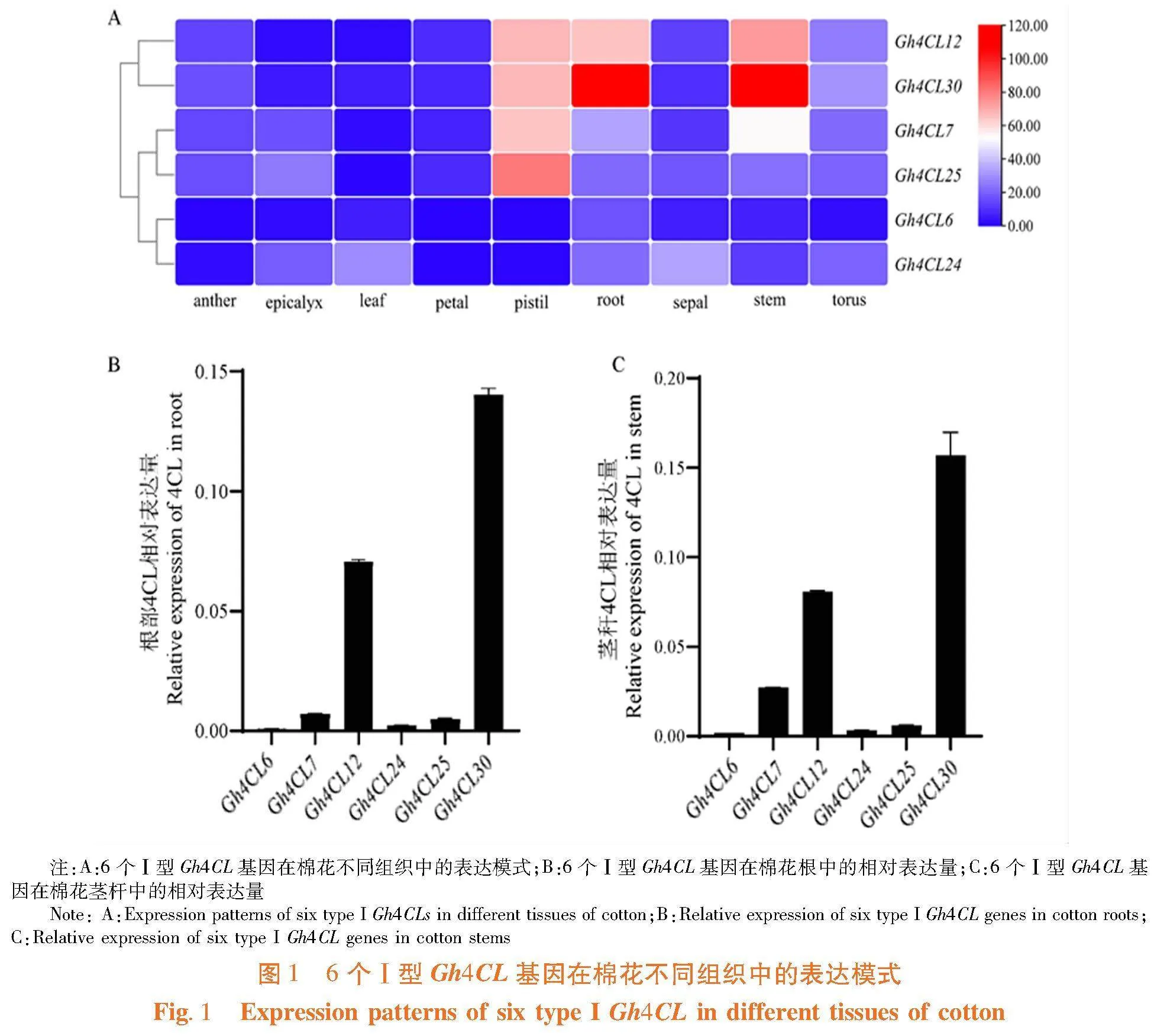
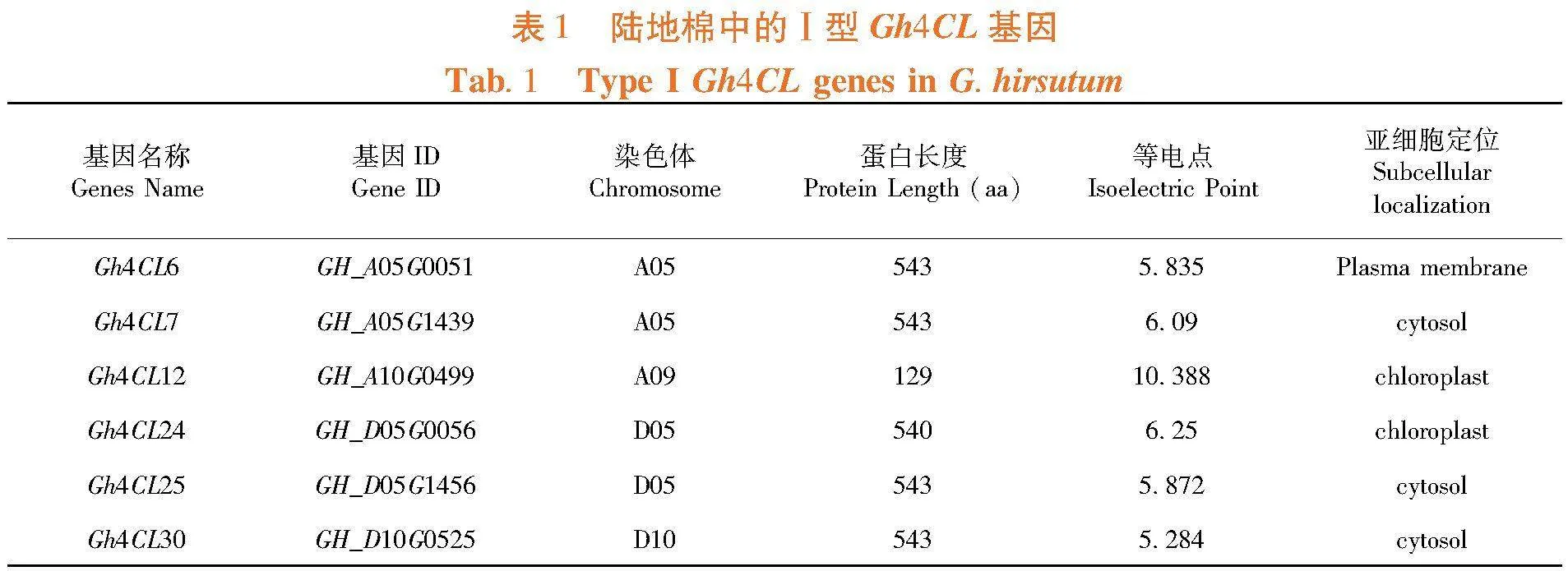
研究表明,鉴定到6个Ⅰ型Gh4CL基因,分布在A05、A09、D05、D10号染色体上,其蛋白长度在129 ~ 543 aa,理论等电点(pI)介于5.28 ~ 10.39,亚细胞定位预测这些基因分别被定位在细胞膜、细胞质和叶绿体中。Gh4CL7和Gh4CL25在雌蕊中优势表达,Gh4CL12和Gh4CL30在雌蕊、根部和茎秆中表达较高,Gh4CL30基因在棉花根和茎中优势表达(图1A)。Gh4CL30基因在根部和茎秆中的表达模式与公共表达谱数据结果一致(图1B,1C)。棉花中Ⅰ型Gh4CL基因具有功能分化,Gh4CL30基因可能在棉花茎秆和根的生长中发挥关键作用。表1,图1
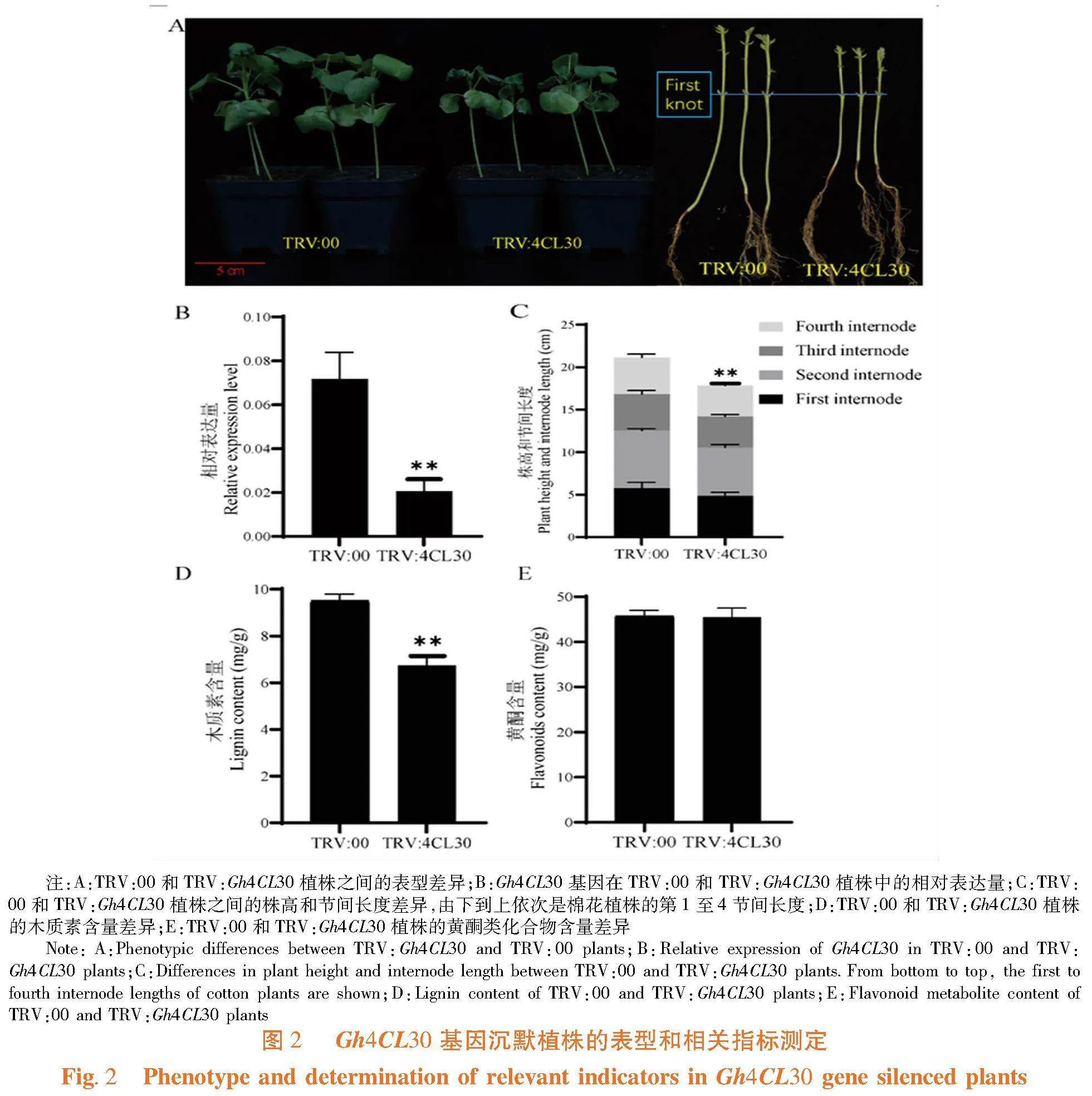
2.2 沉默Gh4CL30基因对棉花木质素含量及株高的影响
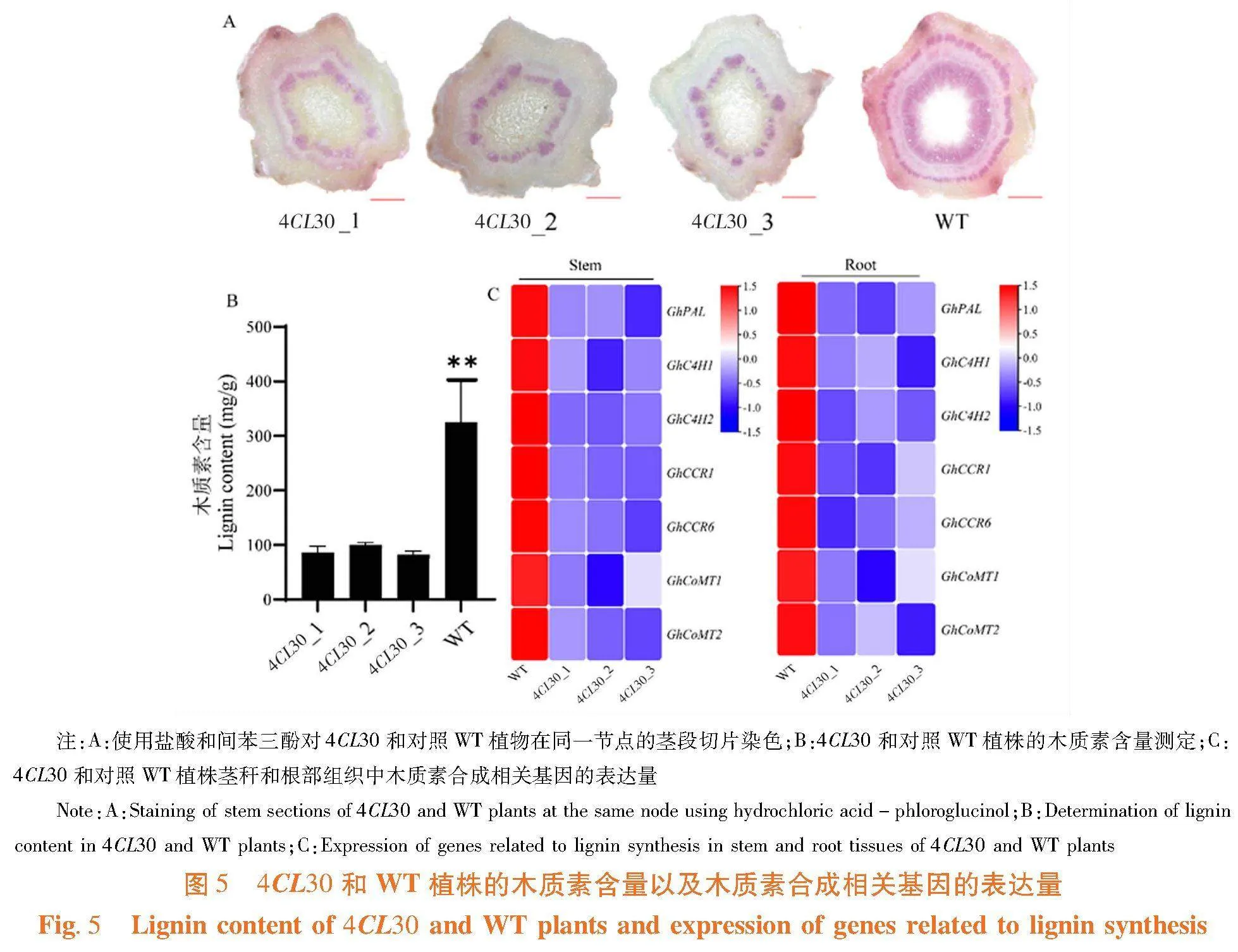
研究表明,沉默植株Gh4CL30基因表达量显著降低(图2B)。与TRV:00相比,TRV:Gh4CL30的整体株高显著降低,降低了约15%(图2A)。此外,TRV:Gh4CL30的前四个节间的长度显著低于TRV:00(图2C)。与对照相比,TRV:Gh4CL30植株茎秆中木质素含量显著降低约29%(图2D),黄酮类化合物含量无显著差异(图2E)。Gh4CL30基因表达被抑制会使棉花木质素生物合成显著下降进而导致植株矮化。图2
2.3 Gh4CL30基因编辑对棉花木质素含量及株高的影响
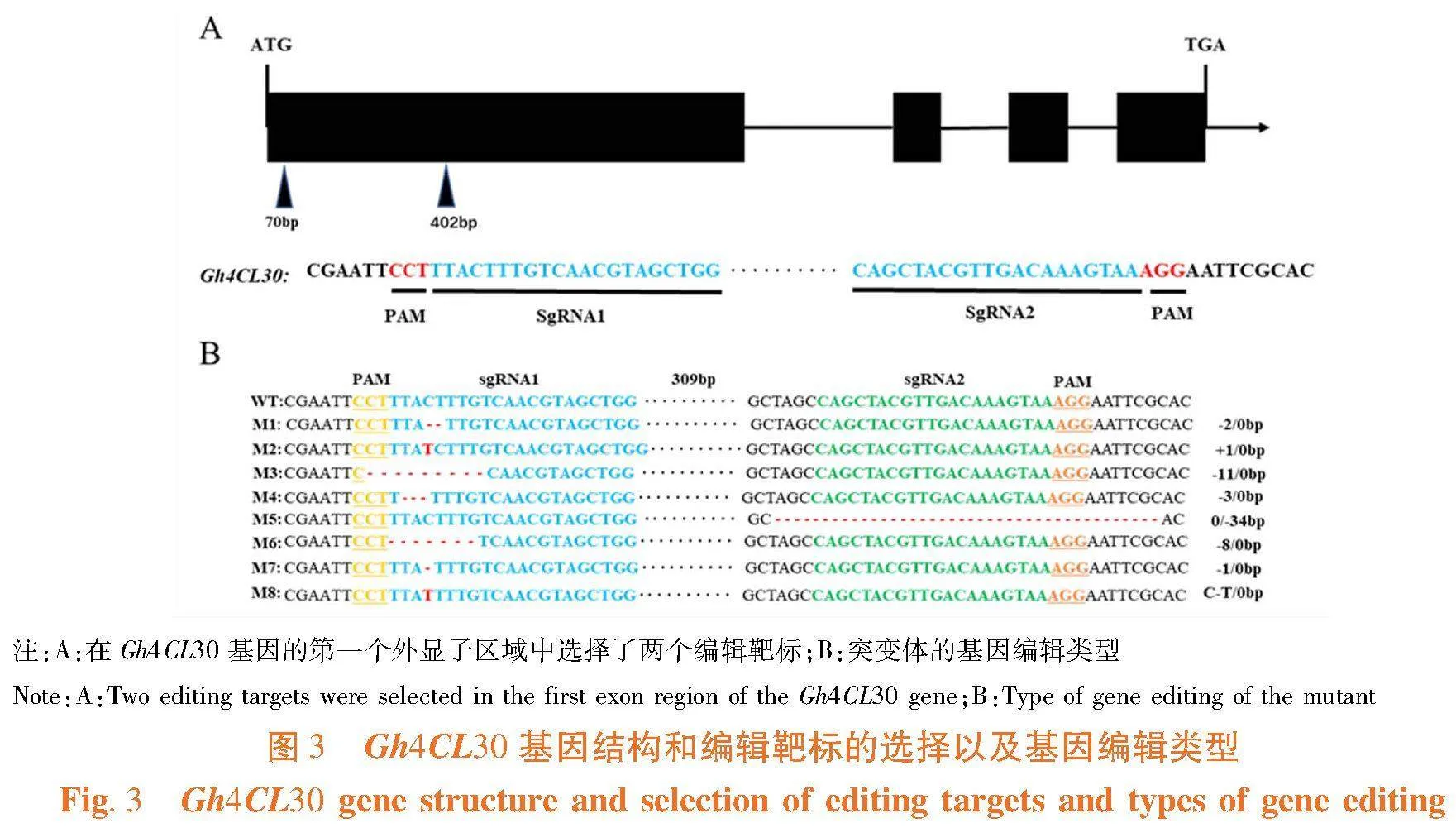
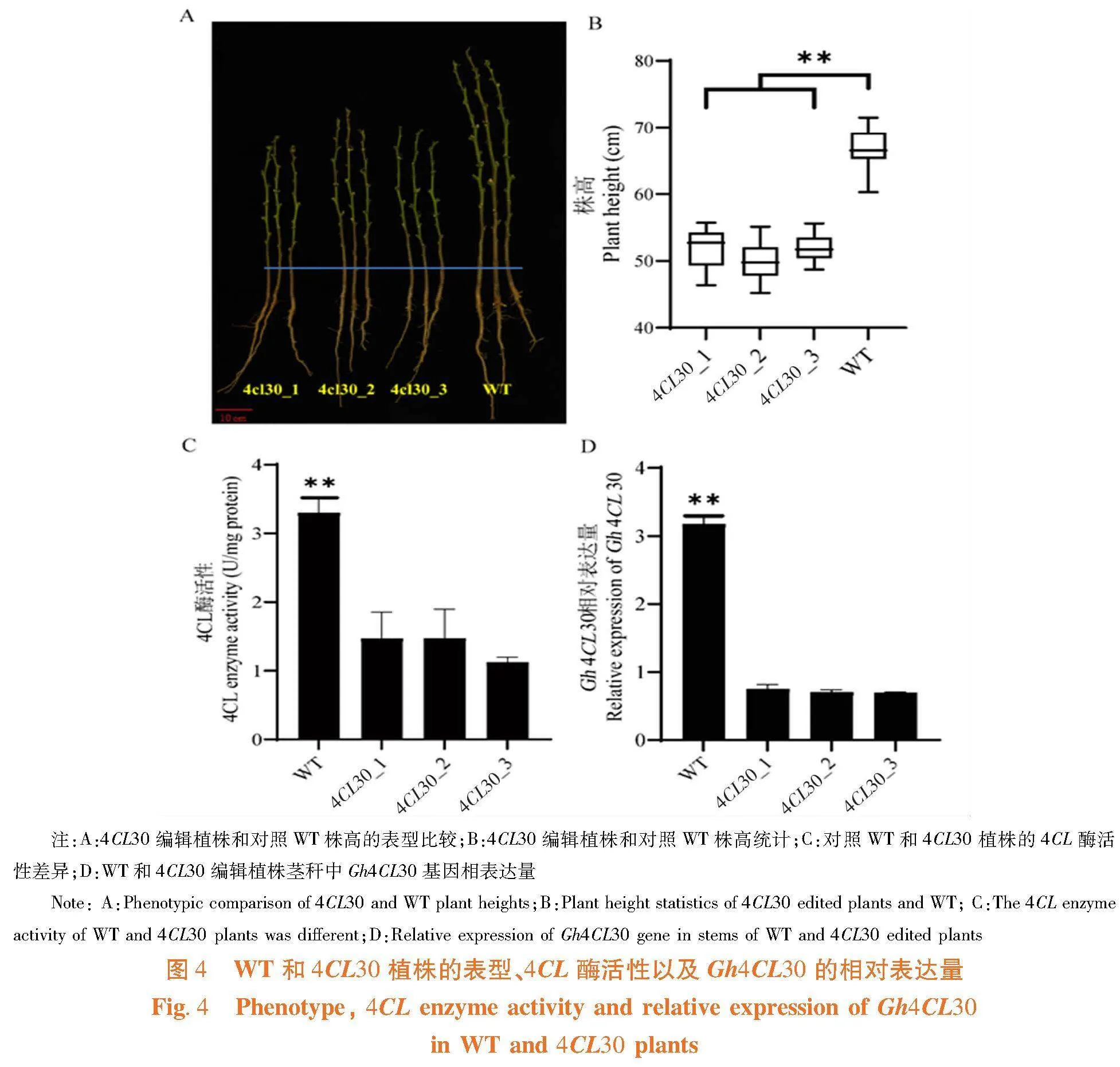
研究表明,针对Gh4CL30基因第一个外显子区段设计sgRNA(图3A)。遗传转化棉花并对阳性植株进行编辑位点验证,共得到8个具有不同编辑类型的独立遗传转化株系(图3B)。图3
与对照WT相比,4CL30基因编辑植株中Gh4CL30转录本显著降低(图4D),4CL酶含量显著下降(图4C),Gh4CL30基因被敲除。与对照WT相比,4CL30基因编辑植株株高降低了约20%,达到显著水平。图3~4
4CL30茎秆中的木质素含量显著低于对照WT(图5B)。4CL30编辑植株茎秆的着色程度明显低于对照WT(图5A),与木质素含量测定结果一致。和对照WT相比,木质素生物合成相关基因在4CL30编辑植株的茎秆和根部中的表达显著降低(图5C),4CL30基因编辑后降低了棉花茎秆中木质素的生物合成。图5
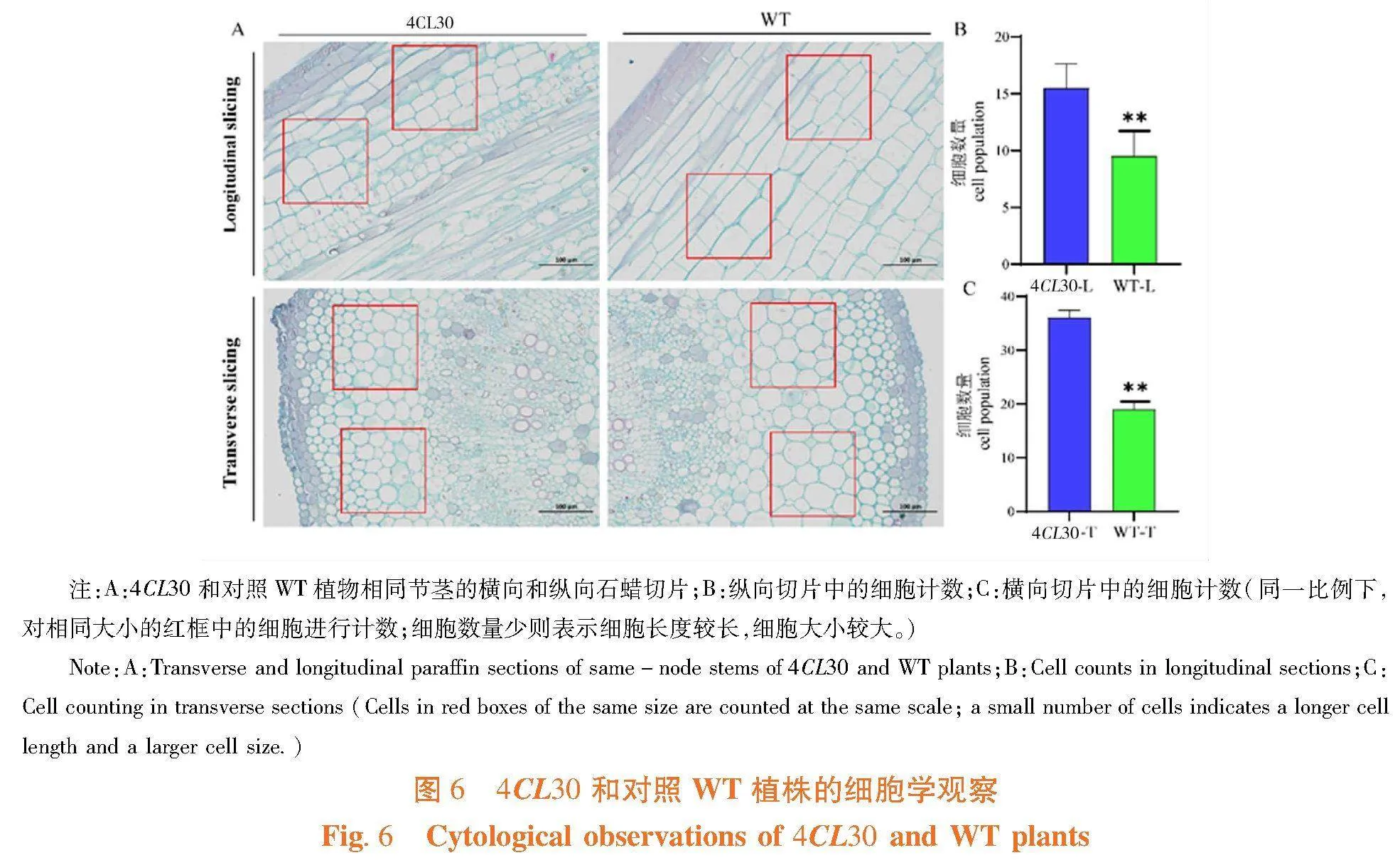
取对照WT和4CL30相同节间的茎秆,进行纵向和横向切片观察,在纵向切片中,与对照WT相比,4CL30编辑植株的皮层细胞明显较短(图6A,6B),在横向切片中,4CL30的皮层细胞显著变小(图6A,6C)。Gh4CL30基因敲除导致棉花茎秆的木质素含量显著降低,引起皮层细胞发育减弱,细胞长度和大小明显小于野生型,从而表现出植株矮化。图6
2.4 Gh4CL30基因敲除导致棉花纤维品质降低
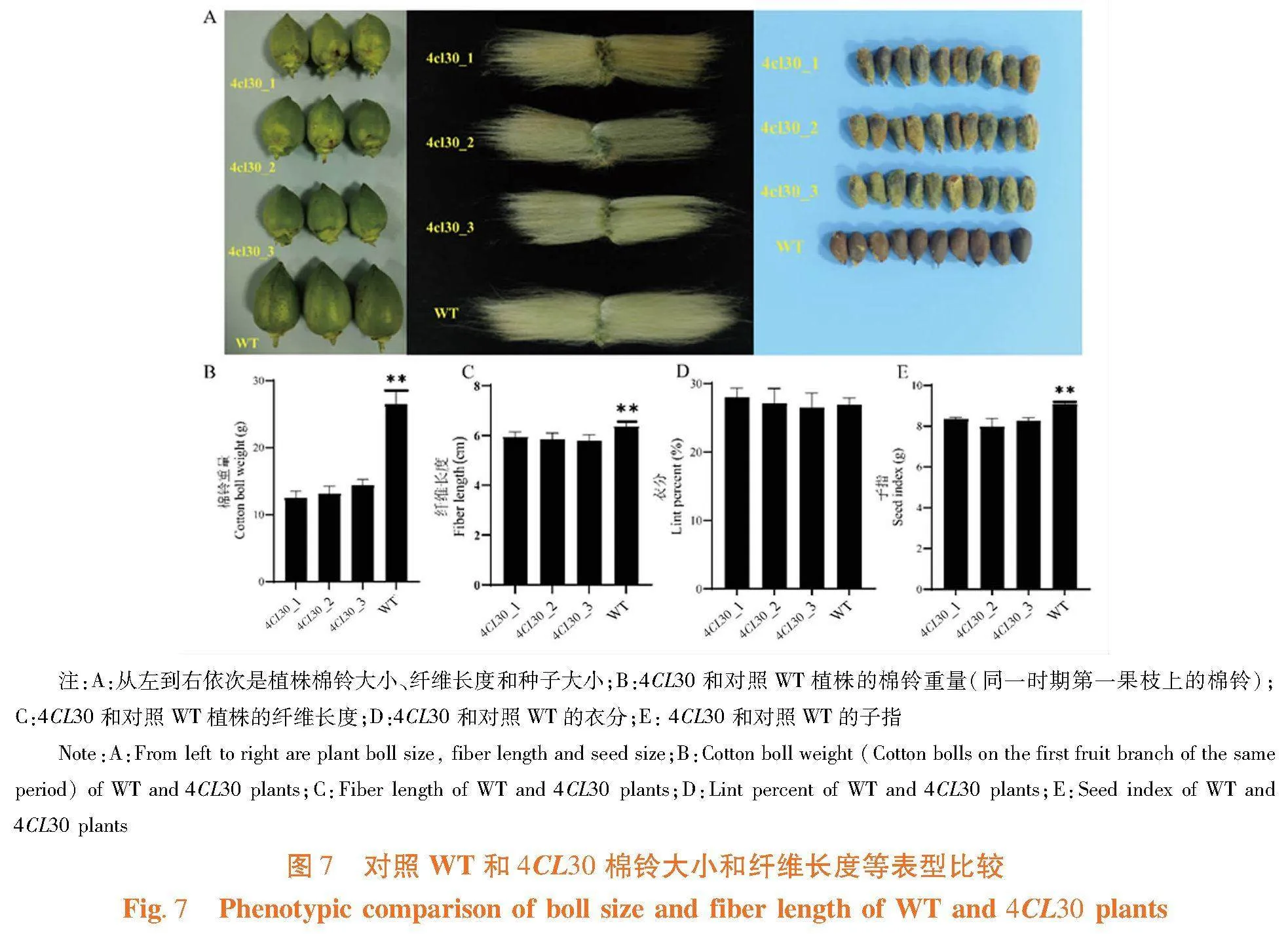
研究表明,4CL30纤维长度显著低于对照WT(图7A,7C)。衣分无显著差异(图7D),子指显著降低(图7E)。除此之外,4CL30敲除植株的棉铃和种子体积变小(图7A,7B)。Gh4CL30基因被敲除后,降低了棉花木质素生物合成,进而影响了纤维品质。图7
3 讨 论
3.1
木质素对棉花纤维品质的形成起着关键作用[24]。
挖掘棉花株高关键调控基因,通过分子手段控制棉花株高,选育出株高适当、株型理想的品种,是棉花育种的一项重要目标[25]。植物株高发育调控是一个复杂的进程。研究表明,木质素含量变化,能够影响植物株高[26]。
3.2
苯丙烷途径为木质素生物合成提供前体物质,4-香豆酸辅酶A连接酶(4CL)作为苯丙烷途径最后的关键酶,对木质素生物合成至关重要[11]。研究中分析了陆地棉中的Ⅰ型Gh4CL基因,共鉴定得到6个Ⅰ型Gh4CL基因,利用公共表达谱数据和荧光定量分析了这些基因在棉花中不同组织中的表达模式,发现Gh4CL30在棉花的根和茎中优势表达。在植物茎的木质部中显著优势表达的4CL基因通常属于Ⅰ型4CL,主要参与木质素的生物合成。白杨中,Ⅰ型4CL基因Pt4CL1和Pt4CL2主要在木质部优势表达并参与木质素的生物合成[23-29]。因此,Gh4CL30基因可能调控陆地棉茎秆中木质素的生物合成。试验研究在棉花中沉默Gh4CL30基因导致棉花木质素含量显著降低,并且导致植株矮化,有研究表明,在杨树中下调4CL基因的表达会使木质素含量降低,引起茎秆矮化以及茎秆强度降低[12]。Gh4CL30基因可能通过调控棉花茎秆中木质素生物合成从而调控株高。
3.3
植物细胞壁的生长需要木质素提供机械支撑,并在整个生命周期中负责运输水分和营养物质,是陆生植物能够长高的必要条件[6]。此外,植物细胞扩增需要细胞壁的连续沉积和修饰[30]。Gh4CL30基因敲除植株(4CL30)株高显著降低,茎秆中木质素含量减少。4CL30茎秆皮层细胞的长度和大小显著小于对照WT。有过表达Fm4CL的转基因烟草中,和野生型相比,转基因品系的木质部细胞层增加40%,木质部细胞壁增加21.6%[12],4CL基因在木质部细胞生长发育过程中发挥重要作用。Gh4CL30基因可能通过影响皮层细胞的细胞壁生成、细胞长度和大小,从而调控株高。
3.4
木质素作为填充棉花纤维素骨架的主要物质,对棉花纤维品质的形成起着关键的作用[24]。在次生壁增厚期,与木质素合成相关的酶家族基因发挥重要作用[31]。Gh4CL30基因被敲除后纤维品质下降,棉桃和种子大小显著降低,Gh4CL30可能通过调控木质素合成影响纤维品质和产量,但相关调控机理还有待进一步研究。
4 结 论
陆地棉Ⅰ型Gh4CL基因中的Gh4CL30基因沉默和敲除均导致棉花植株木质素生物合成显著降低,引起植株矮化,基因敲除还会导致纤维长度下降。Gh4CL30是调节陆地棉木质素生物合成的关键基因,对棉花的株高起到重要调控作用。
参考文献(References)
[1]Sun Q, Xie Y H, Li H M, et al. Cotton GhBRC1 regulates branching, flowering, and growth by integrating multiple hormone pathways[J]. The Crop Journal, 2022, 10(1): 75-87.
[2] Bensen R J, Johal G S, Crane V C, et al. Cloning and characterization of the maize An1 gene[J]. The Plant Cell, 1995, 7(1): 75-84.
[3] Fu J Y, Ren F, Lu X, et al. A tandem array of ent-kaurene synthases in maize with roles in gibberellin and more specialized metabolism[J]. Plant Physiology, "2016, 170(2): 742-751.
[4] Vogt T. Phenylpropanoid biosynthesis[J]. Molecular Plant, "2010, 3(1): 2-20.
[5] Wang S C, Alseekh S, Fernie A R, et al. The structure and function of major plant metabolite modifications[J]. Molecular Plant, 2019, 12(7): 899-919.
[6] Zhao Q. Lignification: flexibility, biosynthesis and regulation[J]. Trends in Plant Science, 2016, 21(8): 713-721.
[7] Nakabayashi R, Saito K. Integrated metabolomics for abiotic stress responses in plants[J]. Current Opinion in Plant Biology, "2015, 24: 10-16.
[8] Le Roy J, Huss B, Creach A, et al. Glycosylation is a major regulator of phenylpropanoid availability and biological activity in plants[J]. Frontiers in Plant Science, "2016, 7: 735.
[9] Amrhein N, Frank G, Lemm G, et al. Inhibition of lignin formation by L-alpha-aminooxy-beta-phenylpropionic acid, an inhibitor of phenylalanine ammonia-lyase[J]. European Journal of Cell Biology, 1983, 29(2): 139-144.
[10] Smart C C, Amrhein N. The influence of lignification on the development of vascular tissue inVigna radiata L[J]. Protoplasma, 1985, 124(1): 87-95.
[11] Lavhale S G, Kalunke R M, Giri A P. Structural, functional and evolutionary diversity of 4-coumarate-CoA ligase in plants[J]. Planta, 2018, 248(5): 1063-1078.
[12] Voelker S L, Lachenbruch B, Meinzer F C, et al. Reduced wood stiffness and strength, and altered stem form, in young antisense 4CL transgenic poplars with reduced lignin contents[J]. The New Phytologist, 2011, 189(4): 1096-1109.
[13] Chen X H, Wang H T, Li X Y, et al. Molecular cloning and functional analysis of 4-Coumarate: CoA ligase 4(4CL-like 1)from Fraxinus mandshurica and its role in abiotic stress tolerance and cell wall synthesis[J]. BMC Plant Biology, "2019, 19(1): 231.
[14] Li Y, Kim J I, Pysh L, et al. Four isoforms of Arabidopsis 4-coumarate: CoA ligase have overlapping yet distinct roles in phenylpropanoid metabolism[J]. Plant Physiology, "2015, 169(4): 2409-2421.
[15] Gui J S, Shen J H, Li L G. Functional characterization of evolutionarily divergent 4-coumarate: coenzyme a ligases in rice[J]. Plant Physiology, 2011, 157(2): 574-586.
[16] Shi R, Sun Y H, Li Q Z, et al. Towards a systems approach for lignin biosynthesis in Populus trichocarpa: transcript abundance and specificity of the monolignol biosynthetic genes[J]. Plant amp; Cell Physiology, "2010, 51(1): 144-163.
[17] Ehlting J, Büttner D, Wang Q, et al. Three 4-coumarate: coenzyme A ligases in Arabidopsis thaliana represent two evolutionarily divergent classes in angiosperms[J]. The Plant Journal, 1999, 19(1): 9-20.
[18] Wang B, Sun W, Li Q S, et al. Genome-wide identification of phenolic acid biosynthetic genes in Salvia miltiorrhiza[J]. Planta, "2015, 241(3): 711-725.
[19] Sun S C, Xiong X P, Zhang X L, et al. Characterization of the Gh4CL gene family reveals a role of Gh4CL7 in drought tolerance[J]. BMC Plant Biology, 2020, 20(1): 125.
[20] Zhang T Z, Hu Y, Jiang W K, et al. Sequencing of allotetraploid cotton (Gossypium hirsutum L. acc. TM-1) provides a resource for fiber improvement[J]. Nature Biotechnology, 2015, 33(5): 531-537.
[21] Chen C J, Chen H, Zhang Y, et al. TBtools: an integrative toolkit developed for interactive analyses of big biological data[J]. Molecular Plant, 2020, 13(8): 1194-1202.
[22] Gao W, Long L, Zhu L F, et al. Proteomic and virus-induced gene silencing (VIGS) Analyses reveal that gossypol, brassinosteroids, and jasmonic acid contribute to the resistance of cotton to Verticillium dahliae[J]. Molecular amp; Cellular Proteomics, 2013, 12(12): 3690-3703.
[23] Cheng X Q, Zhang X Y, Xue F, et al. Characterization and transcriptome analysis of a dominant genic male sterile cotton mutant[J]. BMC Plant Biology, 2020, 20(1): 312.
[24] Fan L, Shi W J, Hu W R, et al. Molecular and biochemical evidence for phenylpropanoid synthesis and presence of wall-linked phenolics in cotton fibers[J]. Journal of Integrative Plant Biology, 2009, 51(7): 626-637.
[25] 付远志, 薛惠云, 胡根海, 等. 我国棉花株型性状遗传育种研究进展[J]. 江苏农业科学, 2019, 47(5): 16-19.
FU Yuanzhi, XUE Huiyun ,HU Genhai, et al. Research progress on genetics breeding of plant architecture traits of cotton in China[J]. Jiangsu Agricultural Sciences, 2019, 47(5): 16-19.
[26] Li X, Bonawitz N D, Weng J K, et al. The growth reduction associated with repressed lignin biosynthesis in Arabidopsis thaliana is independent of flavonoids[J]. The Plant Cell, "2010, 22(5): 1620-1632.
[27] Hu W J, Kawaoka A, Tsai C J, et al. Compartmentalized expression of two structurally and functionally distinct 4-coumarate: CoA ligase genes in aspen (Populus tremuloides)[J]. Proceedings of the National Academy of Sciences of the United States of America, 1998, 95(9): 5407-5412.
[28] Sutela S, Hahl T, Tiimonen H, et al. Phenolic compounds and expression of 4CL genes in silver birch clones and Pt4CL1a lines[J]. PLoS One, "2014, 9(12): e114434.
[29] Allina S M, Pri-Hadash A, Theilmann D A, et al. 4-Coumarate: coenzyme A ligase in hybrid poplar. Properties of native enzymes, cDNA cloning, and analysis of recombinant enzymes[J]. Plant Physiology, "1998, 116(2): 743-754.
[30] Zhong R Q, Cui D T, Ye Z H. Secondary cell wall biosynthesis[J]. The New Phytologist, 2019, 221(4): 1703-1723.
[31] Gou J Y, Wang L J, Chen S P, et al. Gene expression and metabolite profiles of cotton fiber during cell elongation and secondary cell wall synthesis[J]. Cell Research, 2007, 17(5): 422-434.
Functional analysis of 4-coumarate: CoA ligase gene Gh4CL30 in upland cotton
Abstract:【Objective】 To preliminarily study the biological function of 4-coumarate-CoA ligase (4CL) family gene gh4cl30, which might provide theoretical basis and genetic resources for cotton plant breeding.
【Methods】 Virus-induced gene silencing technology and gene editing technology was used to obtain gh4cl30-silenced and edited plants and determine the flavone and lignin contents of the gene suppressed and knockout plants to investigate field phenotype, seed size and fiber quality.
【Results】 "Both gene silencing and gene editing of the gene resulted in dwarfing of the plants and a significant reduction of lignin content in the plant stalks.Field phenotypic measurements showed that seed size and fiber length were significantly reduced in the gene edited plants.
【Conclusion】 "Gh4CL30 affects cotton growth and development mainly through regulating lignin biosynthesis.
Key words:Gossypium hirsutum L.; 4CL; lignin; gene editing; plant height